小分子物质或配体与蛋白质结合的区域被称为结合位点。配体与蛋白质结合后,通常会引起蛋白质结构和功能的变化,进而发挥其特定功能。蛋白质结合位点通常以沟槽的形式存在于蛋白质中,方便小分支物质或配体与之结合,从而改变蛋白质的特性和生物学功能。因此,准确预测结合位点是药物设计或蛋白设计的前提。研究人员提出了多种结合位点预测方法[1]。传统的预测方法通常可分为3类:基于几何形状的方法、基于模板的方法和基于能量的方法。基于几何形状的方法通过蛋白质表面的几何形状信息来预测结合位点。大多数配体结合发生在蛋白质表面的小空腔中,这些空腔对小分子具有较高的亲和力。因此,该方法通过在蛋白质结构中放置探针来探测蛋白质表面的几何特征,从而定位可能的结合空腔。常见的基于几何形状的预测方法包括Fpocket[2]、CASTp[3]、PROSITE[4]、P2Rank[5]和siteFiNDER[6]等。其中Fpocket采用基于Voronoi镶嵌的几何方法,计算速度较快,适合用于大规模筛选蛋白质口袋。基于模板的方法通过在数据库中搜索与查询蛋白质相似的已知蛋白质,并将其结合位点映射到查询蛋白质上。FINDSITE[7]是这些早期方法之一,通过将目标蛋白质与已知的、在进化上相距较近的蛋白质进行对比,确定它们之间的进化关系,并分析其结合位点。由于进化过程中保留下来的结合位点往往具有较强的保守性,因此这些稳定的结合位点可能是目标蛋白的潜在结合位点。基于能量的方法则通过计算结合位点与配体相互作用所产生的能量变化来预测结合位点。这类方法通常需要考虑蛋白质和配体的原子坐标、电子密度分布等信息。典型的基于能量的方法包括FoldX[8]、AutoSite[9]和Glide[10]等。这些方法通过定义能量函数评估蛋白质与配体之间的相互作用。例如,可以使用范德华力、静电相互作用、氢键等多种能量项的组合进行打分,并对蛋白质表面的不同区域进行评估,最终选择能量最低的区域作为预测的结合位点。
近年来,机器学习[11]和深度学习[12]技术在生物信息领域的应用为蛋白质结合位点的预测提供了新的思路。针对借鉴计算机视觉任务的思路,目前典型做法是把蛋白质结构体素化为多通道的3D网格,将蛋白质结合位点预测任务转化为计算机视觉中的目标检测问题。这种方法主要使用U-Net[13]网络架构,包括3个部分,分别是编码器、解码器和跳跃连接,旨在解决2D医学影像分割问题。3D U-Net[14]网络则是将U-Net模型扩展到三维数据的自然延伸。3D U-Net保留了U-Net的核心特征,其中编码器用于逐步提取特征,解码器通过逐步上采样恢复空间分辨率,跳跃连接则负责将编码器部分中的高分辨率特征图传递到解码器部分,以保留图像中的细节和结构信息。
Jiménez等[15]提出一种基于深度卷积神经网络预测方法DeepSite,采用四层卷积神经网络作为特征提取器,最终通过Sigmoid函数输出预测的子网格接近结合口袋的概率。该方法首先将蛋白质体素化为多通道的三维网格,利用局部子网格作为DeepSite的输入,计算出相关的评分。Mylonas等[16]提出一种基于蛋白质表面采样点的局部体素网格方法DeepSurf。每个体素网格接收其内部原子的特征信息,用于进一步分析结合位点。与DeepSite相似,DeepSurf将蛋白质局部体素化为模型的输入,但两者都未能充分利用蛋白质3D结构中的全部信息。Stepniewska-Dziubinska等[17]首次使用3D U-Net结构来预测蛋白质结合位点,该方法将整个蛋白质的三维信息作为模型输入,通过将蛋白质体素化为三维网格,预测三维空间中每个点作为结合位点的概率。Aggarwal等[18]在此基础上,利用Fpocket[2]生成每个预测位点的候选中心,并将以每个候选中心为中心的子网格输入3D U-Net网络中进行进一步分析。Li等[19]提出了一种具有门控循环优化的Lmser网络,设计了从解码器到编码器的前向跳跃连接以及反向的反馈连接,以增强模型的表达能力。Li等[20]则在Lmser网络的基础上引入了注意力机制,通过在小尺度上建立特征依赖关系,进一步细化了对局部区域的结合位点预测。Lin等[21]通过残差结构增强注意力以及自引导掩码模块的集成来捕获蛋白质的多尺度特征。
虽然以上模型在蛋白质结合位点预测中已经取得较好的效果,但是蛋白质的立体结构中含有较多与结合位点无关的信息。在这种复杂的结构中精确预测结合位点仍会受到无关信息或者噪声的干扰。另外,由于结合位点的形状通常无规则,以上模型使用蛋白质单一尺度信息难以精确地捕捉到蛋白质的边界特征。为了解决以上问题,本文提出一种融合注意力机制和多尺度信息的蛋白质结合位点预测模型AMPocket。主要创新点如下。
(1)设计了压缩注意力模块。在编码器和解码器部分均加入了SA(squeezed attention)模块。这一机制使得模型能够更加精确地捕捉到蛋白质中关键的通道信息,减少冗余或无关特征的干扰。进一步增强了对蛋白质特征的利用效率与表达能力。
(2)设计了多尺度信息模块。在编码器中引入了MI(multi-scale information)模块。这一设计使得网络不仅能够从全局特征中提取信息,还能有效地从局部特征中汲取细节,从而提升解码器在特征表示上的能力,获得更加丰富和高层次的特征表达。
1 本文模型
AMPocket模型结构以3D U-Net为骨架,包括编码器部分、解码器部分、跳跃连接部分。如图1所示,模型共包含10个卷积模块,分别为1个Pre块、4个编码器(E1、E2、E3、E4)、4个解码器(D1、D2、D3、D4)和1个Post块。Pre块负责对输入数据进行预处理;编码器提取特征并下采样减小特征图的尺寸;解码器负责逐步恢复特征图的尺寸;编码器与解码器之间通过跳跃连接融合特征;Post块负责输出结果。
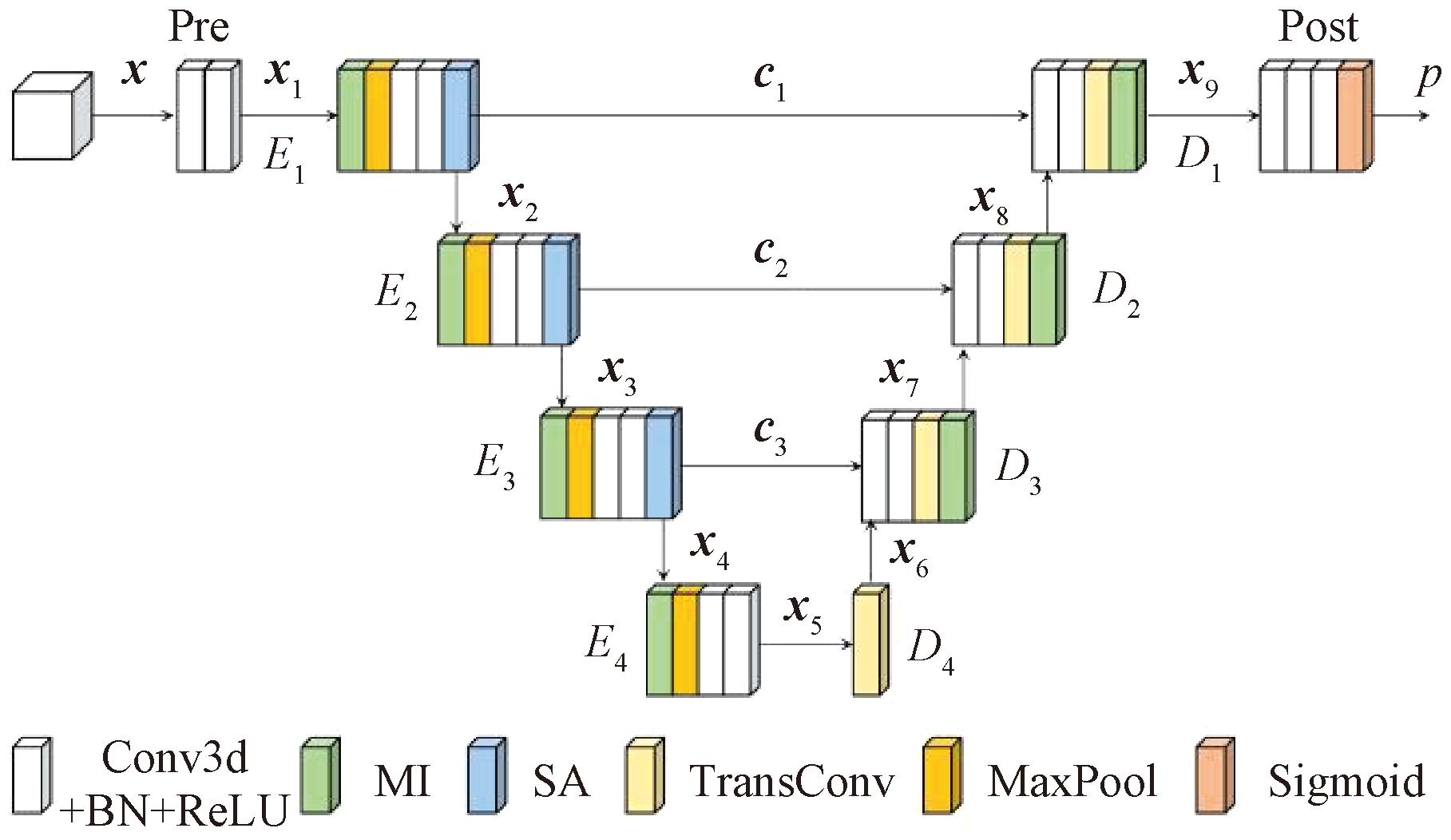
图1 AMPocket模型架构图
Figure 1 AMPocket model architecture
1.1 编码器
对于给定的一个已经体素化的蛋白质数据x,本文首先通过Pre块提取蛋白质的初步特征,为后续编码器部分的处理提供基础。随后使用E1块进一步提取蛋白质的深层特征,并在跳跃连接的过程中应用MI模块,向解码器有效传递信息。接着,继续使用E2、E3、E4提取更多特征,逐步丰富特征表示。整个处理过程如下所示:
x1=Pre(x);
(1)
xi=Ei-1(xi-1),6>i>1;
(2)
ci=Ei(xi),4>i>0。
(3)
1.2 压缩注意力模块
在基于神经网络的分割任务中,SA模块的结构与二维的SE[22]模块类似,但SA模块采用的是整体三维结构,其网络结构如图2所示。
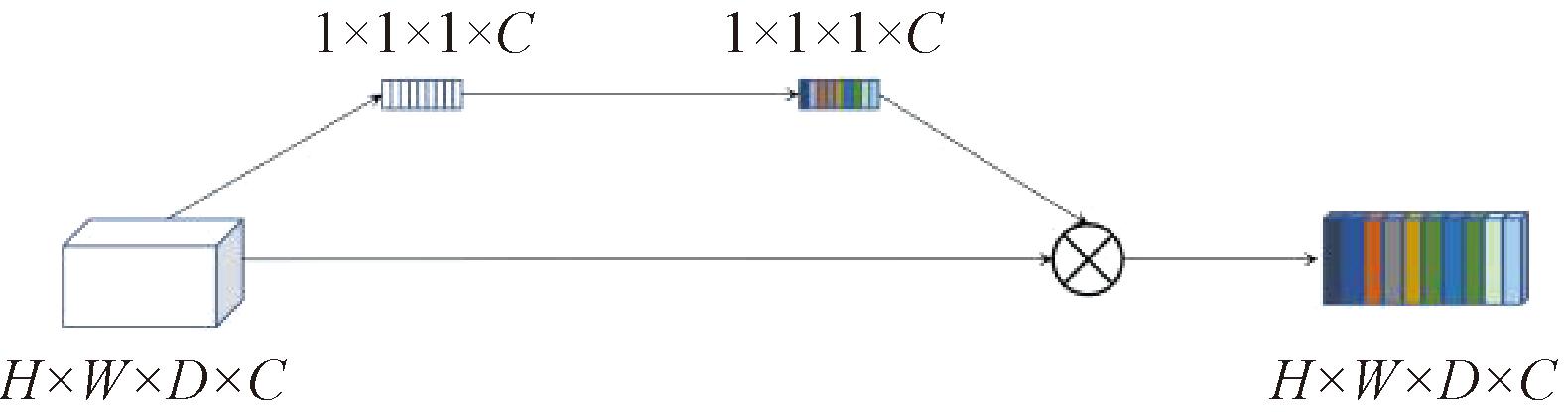
图2 SA模块架构图
Figure 2 SA module architecture
SA通过三维平均池化压缩特征图的维度,生成通道描述符,使用两层全连接结构学习通道间的非线性关系,通过Sigmoid函数生成通道的注意力权重,通过特征重标定生成不同权重的通道。本文采用三维自适应平均池化层来替代SE[22]中的全局平均池化层。全局平均池化会将整个特征图压缩为一个单一值,这种方法在处理三维结构时容易丧失关键的空间信息;而三维自适应平均池化能够有效地捕捉并保留空间结构信息,从而避免了这一问题。
SA模块通过自适应调整每个通道的权重,学习通道间的相互关系,从而改变各个通道的重要性。利用全局信息,SA模块能够识别任务中更加关键的通道,并通过重标定强化这些通道的特征,使得模型能够更聚焦于有价值的关键信息。通过这种方式,SA模块不仅能够提升特征表示能力,还可以增强3D U-Net在复杂图像分割任务中的表现,使其能够更好地捕捉关键通道特征,同时抑制不重要通道特征的影响,从而提高模型的适应性和鲁棒性,提高预测精度。SA模块中的数据处理过程如下所示:
(4)
(5)
1.3 多尺度信息模块
3D U-Net通常仅能获取单一尺度的特征,这可能导致全局或局部信息的丢失。为了应对这一问题,本文提出了多尺度特征融合的三维残差卷积模块(MI),该模块包含多分支特征提取,并将其与残差连接相结合,详细结构如图3所示。本文通过4个分支对输入进行卷积操作,以获取多尺度的特征信息。分支1使用1×1×1的卷积核,将输入的通道数压缩至32个通道。该卷积操作减少了计算复杂度,同时也能有效捕捉输入特征。分支2首先通过一个1×1×1的卷积减少通道数,再使用3×3×3卷积提取局部空间特征,最后通过ReLU激活函数增加非线性,得到中等感受野。分支3首先通过1×1×1卷积减少通道数,接着使用2个连续的3×3×3卷积提取更复杂的空间特征。分支4首先进行最大池化提取最显著特征,然后通过1×1×1卷积恢复通道数。池化操作有助于提取更高层次的特征。最后通过1×1×1卷积将多分支特征从高维空间映射回低维空间,以极少参数量实现复杂特征的交互。此模块通过多条不同路径结构增强了特征表示能力,同时残差连接保证了梯度传播的稳定性。MI模块不仅能从编码器中传递多尺度信息,而且通过跳跃连接将多尺度信息融入解码器。因此,MI模块能够实现不同尺度特征的有效融合,使得模型在解码阶段能够同时利用来自全局和局部的信息,增强特征表示的多样性与准确性。
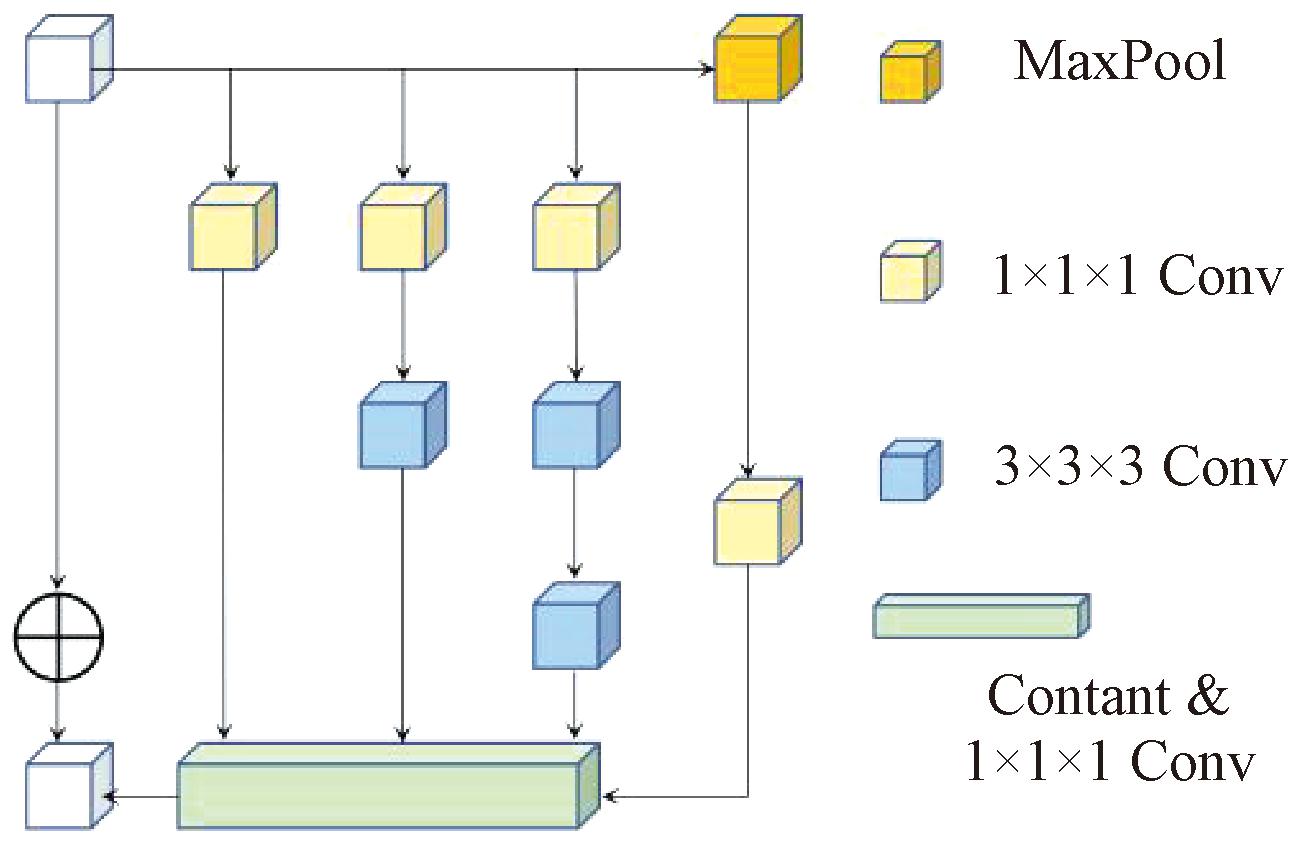
图3 MI模块架构图
Figure 3 MI module architecture
1.4 解码器
解码器模块的主要功能是预测蛋白质的局部结合位点,而非整个蛋白质。这使得其能够更为精确地聚焦于较小的结合区域。解码器的输入包括在前向传播过程中生成的特征表示,以及每一层编码器的输出信息。解码器通过逐步恢复蛋白质的结构尺寸,并结合输入数据,逐渐确定结合口袋的具体位置。最终的预测概率p可通过Post块得到:
p=Post(x9);
(6)
x9=D1(D2(D3(D4(x5),c3),c2,),c1)。
(7)
2 实验
2.1 数据集
本文使用了5个公开数据集来评估AMPocket。具体而言,训练集选用了sc-PDB[23]数据集,测试集则包含COACH420[5]、HOLO4K[5]、PDBbind[24]和SC6K[17]数据集。sc-PDB数据集提供了蛋白质、配体及其结合位点的全原子描述,包括结合位点的几何形状、化学性质等信息,是结合位点预测任务中广泛使用的训练集。该数据集包含16 612个结构、17 594个结合位点和5 540个UniProt ID。本文将该数据集划分为10个部分,使用其中的一部分作为验证集。COACH420和HOLO4K这两个数据集首次在P2Rank[5]方法中用于测试模型性能。SC6K数据集在DeepPocket方法中被提出,包含3 378个蛋白质。本文从PDBbind数据集中剔除25个过大而无法加载的蛋白质,使用剩下的3 888个复合物作为测试集。
在处理数据时,本文使用molgrid库将蛋白质文件体素化,从而获得三维网格数据。
2.2 实验环境及评价指标
AMPocket的训练环境基于PyTorch,批处理大小设置为4,训练时使用NVIDIA GeForce RTX 2080显卡,训练周期为40个epoch,优化器采用SGD,学习率保持在0.001不变,损失函数使用二元交叉熵函数。
本文使用3个指标来评估模型的性能,分别为DCA、DCC和DVO。DCA表示预测口袋中心与最近的配体原子之间的距离,如果距离小于设定的阈值,则认为预测是正确的,否则认为预测是错误的。DCC表示预测口袋中心与标签口袋中心之间的距离,如果DCC小于阈值,则认为预测成功。DVO表示预测口袋与对应标签口袋的重叠部分与它们体积并集的比值。
DCA成功率和DCC成功率表示对应成功预测的口袋数量与总口袋数量的比值。对于DCC指标下的正确预测口袋,本文进一步计算其DVO值;对于未正确预测的口袋,其DVO值被设定为0。
为了确定阈值,Kalasanty等[17]使用0~20Å(间隔1Å)绘制了DCC曲线,结果显示4Å是区分高精度预测与一般预测的临界点。阈值为4Å时达到了覆盖真实位点与减少假阳性的平衡,相关对比工作[16-19]均采用4Å为阈值。因此在本文的实验中,也将阈值设定为4Å。
2.3 对比实验
本文在4个不同的测试集上对AMPocket与最相关对比方法进行了比较,结果如表1所示。其中DeepPocket[18]、RecurPocket[19]、GLPocket[20]和ResPocket[21]是进行蛋白质结合位点预测任务的最新方法。
表1 AMPocket与其他模型在DCC和DVO上对比
Table 1 Comparison of AMPocket with other models on DCC and DVO %
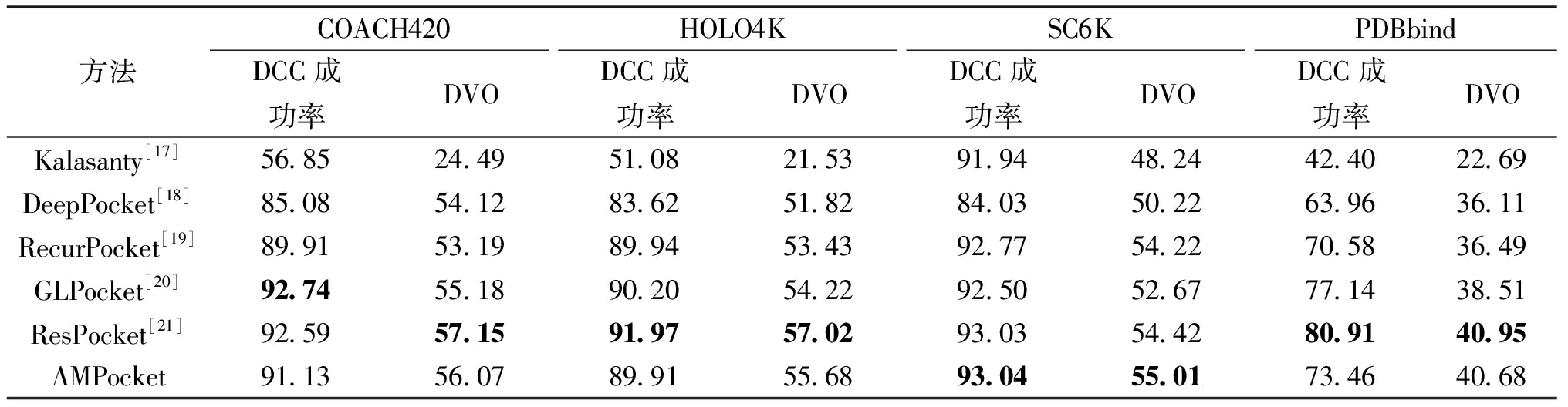
方法COACH420HOLO4KSC6KPDBbindDCC成功率DVODCC成功率DVODCC成功率DVODCC成功率DVOKalasanty[17]56.8524.4951.0821.5391.9448.2442.4022.69DeepPocket[18]85.0854.1283.6251.8284.0350.2263.9636.11RecurPocket[19]89.9153.1989.9453.4392.7754.2270.5836.49GLPocket[20]92.7455.1890.2054.2292.5052.6777.1438.51ResPocket[21]92.5957.1591.9757.0293.0354.4280.9140.95AMPocket91.1356.0789.9155.6893.0455.0173.4640.68
结果表明,本文提出的AMPocket通过减少噪声干扰和利用蛋白质的多尺度信息,整体上取得了比较优异的表现,特别是在SC6K数据集上的DCC成功率和DVO优于所有对比方法。由于传统卷积难以捕捉局部区域的长距离依赖关系,GLPocket和ResPocket中均添加了Transformer,利用自注意力机制和位置编码,提取了patch之间的相对位置关系,提升了空间结构的感知能力,能更精准地定位口袋中心。但是和最新提出的ResPocket相比,AMPocket在SC6K数据集上仍保持优势。相比之下,AMPocket通过压缩注意力机制和多尺度特征融合,充分利用蛋白质的各种特征信息,缓解了传统3D U-Net在蛋白质结合位点预测中的单一特征问题。
如图4所示,标准的3D U-Net、GLPocket和AMPocket对同一蛋白(ID:1AEF)进行结合位点预测,所得到的DVO分别为43.70%、46.25%和68.63%,AMPocket得到最优结果。
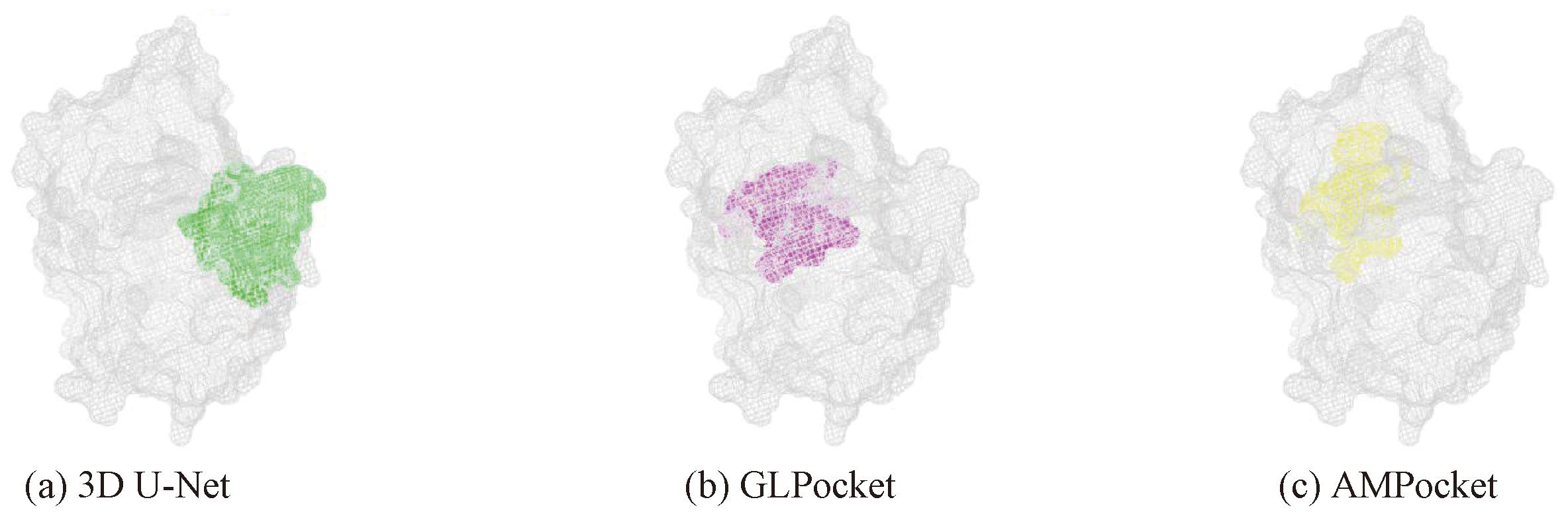
图4 标准的3D U-Net、GLPocket和AMPockt所预测结合位点的结合位置及形状
Figure 4 Location and shape of binding sites predicted by standard 3D U-Net,GLPocket,and AMPocket
如表2所示,本文还使用DCA成功率对分类模型与不同基线模型的性能进行了比较。DCA主要评估模型在找到结合位点位置时的排序能力和定位准确度。Top-n衡量的是对预测结果的排序能力,以及对于目标口袋的定位能力;Top-(n+2)则在此基础上对前n个预测的排名进行扩展。
表2 AMPocket与其他模型在Top-n和Top-(n+2)上的DCA成功率对比
Table 2 Comparison of AMPocket with other models on DCA success rate of Top-n and Top-(n+2)
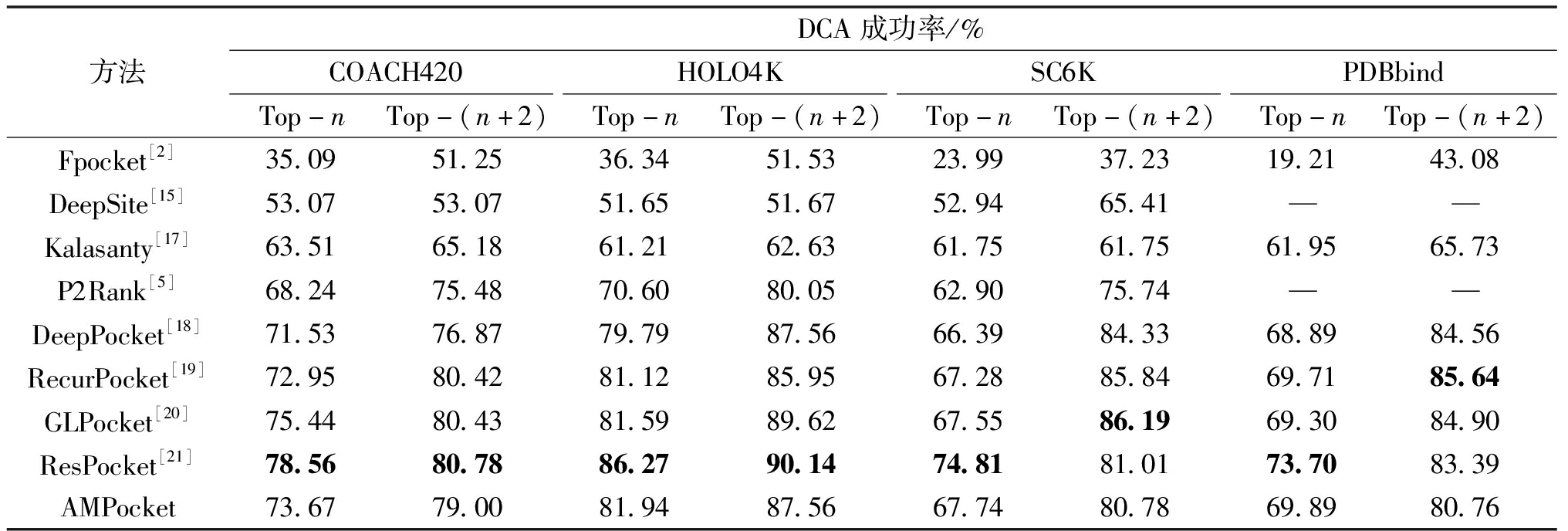
方法DCA成功率/%COACH420HOLO4KSC6KPDBbindTop-nTop-(n+2)Top-nTop-(n+2)Top-nTop-(n+2)Top-nTop-(n+2)Fpocket[2]35.0951.2536.3451.5323.9937.2319.2143.08DeepSite[15]53.0753.0751.6551.6752.9465.41——Kalasanty[17]63.5165.1861.2162.6361.7561.7561.9565.73P2Rank[5]68.2475.4870.6080.0562.9075.74——DeepPocket[18]71.5376.8779.7987.5666.3984.3368.8984.56RecurPocket[19]72.9580.4281.1285.9567.2885.8469.7185.64GLPocket[20]75.4480.4381.5989.6267.5586.1969.3084.90ResPocket[21]78.5680.7886.2790.1474.8181.0173.7083.39AMPocket73.6779.0081.9487.5667.7480.7869.8980.76
由表2可知,AMPocket在4个测试集上的表现优于大多数对比方法,特别是在HOLO4K、SC6K和PDBbind测试集中的前n个口袋的预测仅次于表现最优的ResPocket。虽然与GLPocket和ResPocket相比,AMPocket在某些测试上的表现略逊一筹,但是值得注意的是,AMPocket的参数量仅为GLPocket的95%。
2.4 消融实验
为了分析SA和MI模块的效率,本文进行了消融实验。模型在4个测试数据集上进行评估,结果如表3所示。
表3 消融实验
Table 3 Ablation experiment
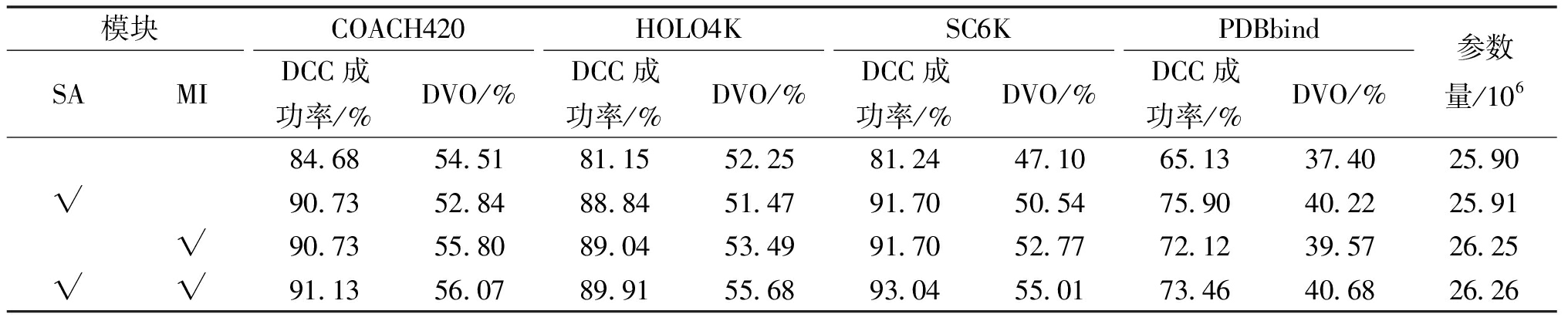
模块COACH420HOLO4KSC6KPDBbindSAMIDCC成功率/%DVO/%DCC成功率/%DVO/%DCC成功率/%DVO/%DCC成功率/%DVO/%参数量/10684.6854.5181.1552.2581.2447.1065.1337.4025.90√90.7352.8488.8451.4791.7050.5475.9040.2225.91√90.7355.8089.0453.4991.7052.7772.1239.5726.25√√91.1356.0789.9155.6893.0455.0173.4640.6826.26
实验结果表明,加入SA模块后,DCC成功率和DVO均表现出显著优势。在4个测试数据集上,DCC成功率提升了6.05百分点~10.77百分点。它使模型能够更加关注重要的蛋白质信息,从而在全局范围内提高了特征提取能力。加入SA模块后的模型参数量相较于3D U-Net的参数量仅仅增加了0.04%,但模型的预测性能得到了显著提升,进一步验证了SA模块的有效性。此外,加入了MI模块后,模型能够更好地捕捉蛋白质的全局和局部特征,从而提高对结合位点的分割精度。实验结果显示,4个测试集上的DVO均显著提高,表明多尺度信息的引入有效增强了模型对结合位点形状的预测准确性。
3 结论
本文提出了一种基于3D U-Net的模型AMPocket,用于捕捉蛋白质的多尺度特征并预测结合位点。在编码器和解码器部分设计了压缩注意力模块,通过学习通道之间的关系自适应调整通道的权重,帮助模型关注更重要的区域,减少了噪声对结合位点预测的干扰。此外,通过在编码器中引入多尺度信息模块,获取到蛋白质的多尺度特征并丰富了全局上下文信息。实验结果表明,在标准3D U-Net模型的基础上仅增加1.4%参数量的情况下,AMPocket在各项指标上均展现出卓越的性能,充分说明了AMPocket在蛋白质结合位点预测任务中的有效性。
[1] XIA Y,PAN X Y,SHEN H B.A comprehensive survey on protein-ligand binding site prediction[J].Current Opinion in Structural Biology,2024,86:102793.
[2] LE GUILLOUX V,SCHMIDTKE P,TUFFERY P.Fpocket:an open source platform for ligand pocket detection[J].BMC Bioinformatics,2009,10:168.
[3] BINKOWSKI T A,NAGHIBZADEH S,LIANG J.CASTp:computed atlas of surface topography of proteins[J].Nucleic Acids Research,2003,31(13):3352-3355.
[4] SIGRIST C J A,DE CASTRO E,CERUTTI L,et al.New and continuing developments at PROSITE[J].Nucleic Acids Research,2013,41:D344-D347.
[5] KRIV K R,HOKSZA D.P2Rank:machine learning based tool for rapid and accurate prediction of ligand binding sites from protein structure[J].Journal of Cheminformatics,2018,10(1):39.
K R,HOKSZA D.P2Rank:machine learning based tool for rapid and accurate prediction of ligand binding sites from protein structure[J].Journal of Cheminformatics,2018,10(1):39.
[6] INNIS C A.siteFiNDER|3D:a web-based tool for predicting the location of functional sites in proteins[J].Nucleic Acids Research,2007,35:W489-W494.
[7] ZHOU H Y,CAO H N,SKOLNICK J.FINDSITEcomb2.0:a new approach for virtual ligand screening of proteins and virtual target screening of biomolecules[J].Journal of Chemical Information and Modeling,2018,58(11):2343-2354.
[8] SCHYMKOWITZ J,BORG J,STRICHER F,et al.The FoldX web server:an online force field[J].Nucleic Acids Research,2005,33:W382-W388.
[9] RAVINDRANATH P A,SANNER M F.AutoSite:an automated approach for pseudo-ligands prediction-from ligand-binding sites identification to predicting key ligand atoms[J].Bioinformatics,2016,32(20):3142-3149.
[10] FRIESNER R A,BANKS J L,MURPHY R B,et al.Glide:a new approach for rapid,accurate docking and scoring.1.method and assessment of docking accuracy[J].Journal of Medicinal Chemistry,2004,47(7):1739-1749.
[11] 池燕飞,李春,冯旭东.机器学习在蛋白质功能预测领域的研究进展[J].生物工程学报,2023,39(6):2141-2157.CHI Y F,LI C,FENG X D.Advances in machine learning for predicting protein functions[J].Chinese Journal of Biotechnology,2023,39(6):2141-2157.
[12] KOZLOVSKII I,POPOV P.Computational methods for binding site prediction on macromolecules[J].Quarterly Reviews of Biophysics,2025,58:1-31.
[13] RONNEBERGER O,FISCHER P,BROX T.U-Net:convolutional networks for biomedical image segmentation[C]∥ Medical Image Computing and Computer Assisted Intervention-MICCAI 2015.Cham:Springer,2015:234-241.
[14] ÇIÇEK Ö,ABDULKADIR A,LIENKAMP S S,et al.3D U-Net:learning dense volumetric segmentation from sparse annotation[J].Lecture Notes in Computer Science,2016,9901:424-432.
[15] JIMÉNEZ J,DOERR S,MART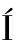 NEZ-ROSELL G,et al.DeepSite:protein-binding site predictor using 3D-convolutional neural networks[J].Bioinformatics,2017,33(19):3036-3042.
NEZ-ROSELL G,et al.DeepSite:protein-binding site predictor using 3D-convolutional neural networks[J].Bioinformatics,2017,33(19):3036-3042.
[16] MYLONAS S K,AXENOPOULOS A,DARAS P.DeepSurf:a surface-based deep learning approach for the prediction of ligand binding sites on proteins[J].Bioinformatics,2021,37(12):1681-1690.
[17] STEPNIEWSKA-DZIUBINSKA M M,ZIELENKIEWICZ P,SIEDLECKI P.Improving detection of protein-ligand binding sites with 3D segmentation[J].Scientific Reports,2020,10(1):5035.
[18] AGGARWAL R,GUPTA A,CHELUR V,et al.DeepPocket:ligand binding site detection and segmentation using 3D convolutional neural networks[J].Journal of Chemical Information and Modeling,2022,62(21):5069-5079.
[19] LI P Y,CAO B H,TU S K,et al.RecurPocket:recurrent Lmser network with gating mechanism for protein binding site detection[C]∥2022 IEEE International Conference on Bioinformatics and Biomedicine (BIBM).Piscataway:IEEE,2022:334-339.
[20] LI P Y,LIU Y C,TU S K,et al.GLPocket:a multi-scale representation learning approach for protein binding site prediction[C]∥ IJCAI International Joint Conference on Artificial Intelligence.Macao:JCAI,2023:4821-4828.
[21] LIN X L,YANG W L,CHEN Y Y,et al.ResPocket:a multi-scale feature fusion method for improving protein binding site detection[C]∥2024 IEEE International Conference on Bioinformatics and Biomedicine (BIBM).Piscataway:IEEE,2024:1588-1591.
[22] HU J,SHEN L,SUN G.Squeeze-and-excitation networks[C]∥2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition.Piscataway:IEEE,2018:7132-7141.
[23] KELLENBERGER E,MULLER P,SCHALON C,et al.sc-PDB:an annotated database of druggable binding sites from the Protein Data Bank[J].Journal of Chemical Information and Modeling,2006,46(2):717-727.
[24] WANG R X,FANG X L,LU Y P,et al.The PDBbind database:methodologies and updates[J].Journal of Medicinal Chemistry,2005,48(12):4111-4119.