阿尔茨海默病(Alzheimer′s disease,AD)是一种不可逆的中枢神经系统衰退性疾病,病程慢性迁延并进行性发展,患者会从正常(healthy control,HC)经过轻度认知障碍(mild cognitive impairment,MCI)发展到AD,最终完全丧失自理能力。其症状主要是渐进性认知功能和行为能力衰退,如记忆力衰减、失语、视空间障碍等。然而,当这些病症出现时大多数患者已经进入病程的中晚期,如果早发现、早干预、早治疗,可在一定程度上减缓阿尔茨海默病的发展速度。因此,如何在大脑形态变化不明显的早期及时进行诊断有着很高的研究价值。
AD早期的主要形态学特征为内侧颞叶的萎缩,尤其是海马体及杏仁核的萎缩,目前主流的AD诊断方法是海马体的影像学评估,精确地勾画海马体能极大地辅助AD等疾病的诊断。磁共振成像(magnetic resonance imaging,MRI)作为一种无创、无辐射的技术,已被广泛应用于影像学检查,是后续评估和诊断疾病的重要依据,而在MRI上手动分割海马体图像需要专业医师来逐层勾画,费时费力。随着计算机技术的不断进步,通过计算机自动分割海马体图像成为研究的热点。
目前,通过FreeSurfer可实现海马体及其他大脑区域图像的自动分割,但该方法需要消耗过多的时间,不具备实时性[1]。近年来,深度学习方法在图像识别领域发展迅速。该方法凭借其网络层较深的特点,可对复杂、多样的特征进行提取,而更高维度、更大规模的神经影像又为基于深度学习方法的计算机自动分割技术提供了良好的素材。因此,深度学习方法在海马体图像分割任务中得到了广泛的应用[2-3]。
虽然深度学习方法在图像分割任务中取得了优异的表现,但由于MRI中的海马体边界不清晰、形状不规则,且不同个体海马体的位置、形状存在一定的差异,使该方法在海马体分割图像任务中的准确性难以提高。此外,处理3D图像需要更多内存开销,且处理过程可能受到冗余信息的干扰[4]。针对这些问题,本文提出了一种二段式的海马体图像分割模型MVF-2.5D U-Net,通过引入通道扩充和多视图融合机制分别对不同视图及上下文信息进行充分提取,旨在进一步提升海马体图像自动分割方法的准确率。
1 相关工作
1.1 分割网络
海马体图像分割方法如图1所示。基于深度学习的海马体图像分割方法主要通过卷积神经网络(convolutional neural networks,CNN)来实现。MRI是由多切面组成的三维影像,3D CNN可以直接将处理后的3D图像作为输入,如图1中(a)所示。
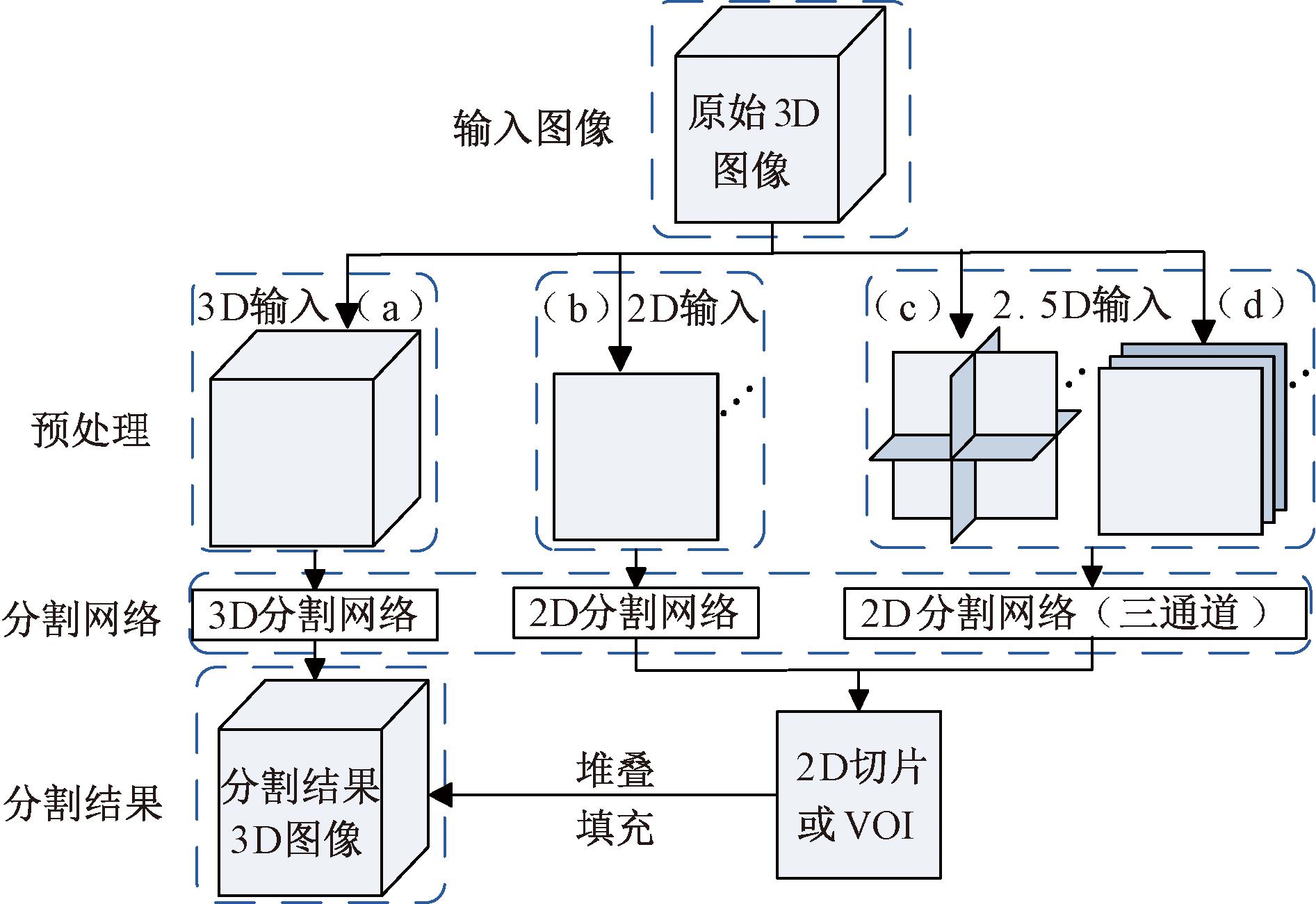
图1 海马体图像分割方法示意图
Figure 1 Schematic diagram of the hippocampus image segmentation method
Liu等[5]通过一个含有残差模块的深度3D CNN网络实现了海马体图像分割,并在其分割结果的基础上通过另一个深度残差网络实现AD与HC的分类。U-Net作为一种对称的U型CNN,由编码器和解码器两部分组成,该网络通过引入跳跃连接(skip connection)解决了编码过程中因最大池化丢失过多图像细节的问题,使得输出图像在保持与输入图像大小相同的前提下依然具有较高的分辨率,适合处理图像分割任务。ÇiÇek等[6]通过扩展2D U-Net提出了3D U-Net模型。Manjon等[7]提出了一个改进的3D U-Net,通过结合模型中不同分辨率的特征图实现了海马体图像分割。然而,直接采用3D卷积网络对数据进行处理的方法虽然能够关注到空间信息,但与2D网络相比,往往会造成较重的内存开销和计算负担,并可能出现信息的冗余,甚至导致特征提取过程受到更多无关特征的干扰[4]。
与3D网络相比,2D网络的优势在于计算量较小,然而,3D图像不能直接输入到2D网络中,需要将图像沿某个视图方向依次处理为一系列的2D切片,如图1(b)所示,随后再将切片逐个输入到2D网络中进行处理,处理完成后,将处理结果按原顺序堆叠并恢复到与原始图像相同的尺寸[8]。Zettler等[9]在肝脏、脾脏、肾脏的图像分割实验中,证明了在2D U-Net中使用水平面的切片比在3D U-Net中使用3D图像准确率更高,原因是这些器官图像形状规则、边界清晰,具有较强的辨识度,仅沿单个视图使用2D网络已经能表现出较好的分割效果。然而,这种方法在分割体积较小或形状不规则的器官图像时表现较差,原因是如果切片方向选择不佳,则会导致切片中该器官的辨识度较差。而单层切片又无法参考上下文信息,针对此问题,许多研究将多个切片组合成3通道的2.5D图像块作为输入。Roth等[10]以兴趣体(volume of interest,VOI)为中心,将3张相互正交的切片组成2.5D三通道图像作为输入,如图1中(c)处所示,以此来检测该VOI处为淋巴结的概率,类似做法还被用在胰腺的分割任务中[11]。这种正交的图像虽然在一定程度上利用了空间信息,但是3个通道间并不存在直接的空间关系,因此该方法具有一定的局限性。Yu等[12]将中心切片与相邻两切片组成3通道的2.5D图像块,如图1中(d)处所示,以此实现小器官的图像分割,这种方法在利用空间信息的同时使图像块保持了与原始图像相同的空间关系,往往能表现出更好的性能。
1.2 注意力机制
CNN具有强大的处理复杂非线性关系的能力,可以处理高维度、大尺寸的数据,但由于卷积核的形状固定、感受野有限,网络需要足够的深度才能覆盖到全局特征。ViT(vision Transformer)的提出使Transformer模型开始广泛应用于计算机视觉领域,其核心在于引入了注意力机制,使网络通过训练可以更多地关注重点区域,具有更强的灵活性。
然而,单独使用Transformer容易造成过拟合,且需要大量的数据进行预训练,越来越多的研究是在CNN网络中嵌入注意力模块。TransUNet[13]将U-Net编码器中的卷积模块替换为Transformer模块以达到更好的特征提取效果,这种结构也常被应用于海马体图像分割任务中[14]。Oktay等[15]提出了用于胰腺图像分割的Attention U-Net模型,其在U-Net的跳跃连接处加入了一个注意门控(attention gate)模块,以调整编码器的输出;Wang等[16]提出了一个类似于3D U-Net网络的U型编码器与解码器架构DeepHipp,并在解码器拼接点之前的跳跃连接处加入了SE(squeeze-and-excitation)模块;魏明军等[17]在SSD中加入了混合注意力模块SeSA以增强特征提取过程中模型对关键信息的捕捉能力,并建立信息间的长距离依赖关系;高宇飞等[18]设计了一种CNN-Transformer模型,同时结合了CNN擅长捕捉局部关系和Transformer擅长捕捉信息间长距离依赖关系的优势,实现了脑胶质瘤图像的分割。这些注意力模块可以轻松地嵌入到网络中,且不破坏网络的原有结构,通过与CNN结合可在不增加过多参数的前提下提高图像分割的准确率。
1.3 多网络融合模型
单个CNN在3D图像识别任务中的准确率通常是有限的,许多研究使用了多网络融合的方法来结合不同网络的优点。Zhou等[19]通过两组模型的串联实现了胰腺图像分割,第一组模型用于VOI的选择,第二组模型用于精分割,每组模型均在矢状面、冠状面、水平面3个方向上通过3个2D U-Net对切片进行处理。Yu等[12]也在3个方向上采用了一种粗分割和精分割结合的两阶段网络实现了小器官图像的分割。这两项研究均采取了众数投票机制对3个视图方向上的分割结果进行融合。Xia等[20]则建立了一个体积融合网络(volumetric fusion net,VFN)来融合3个子网络的分割结果。网络的融合方式在很大程度上影响了信息的利用率和分割结果的准确率。
2 MVF-2.5D U-Net方法介绍
本文提出了一种基于多视图融合和2.5D U-Net的海马体图像分割模型(muti-view fusion and 2.5D U-Net,MVF-2.5D U-Net)。模型主要分为两个阶段,由于多视图切片可以提供更多的信息,模型在矢状面、冠状面、水平面3个方向上均采用了本文改进的2.5D U-Net来实现第一阶段的分割。U-Net在图像分割领域具有良好的性能,并且能保持输出图像和输入图像的尺寸完全相同。模型第二阶段通过本文提出的体积融合网络将3个子网络的分割结果进行融合,以决定每个体素点最终的类别,具体的网络结构如图2所示,处理过程如下:
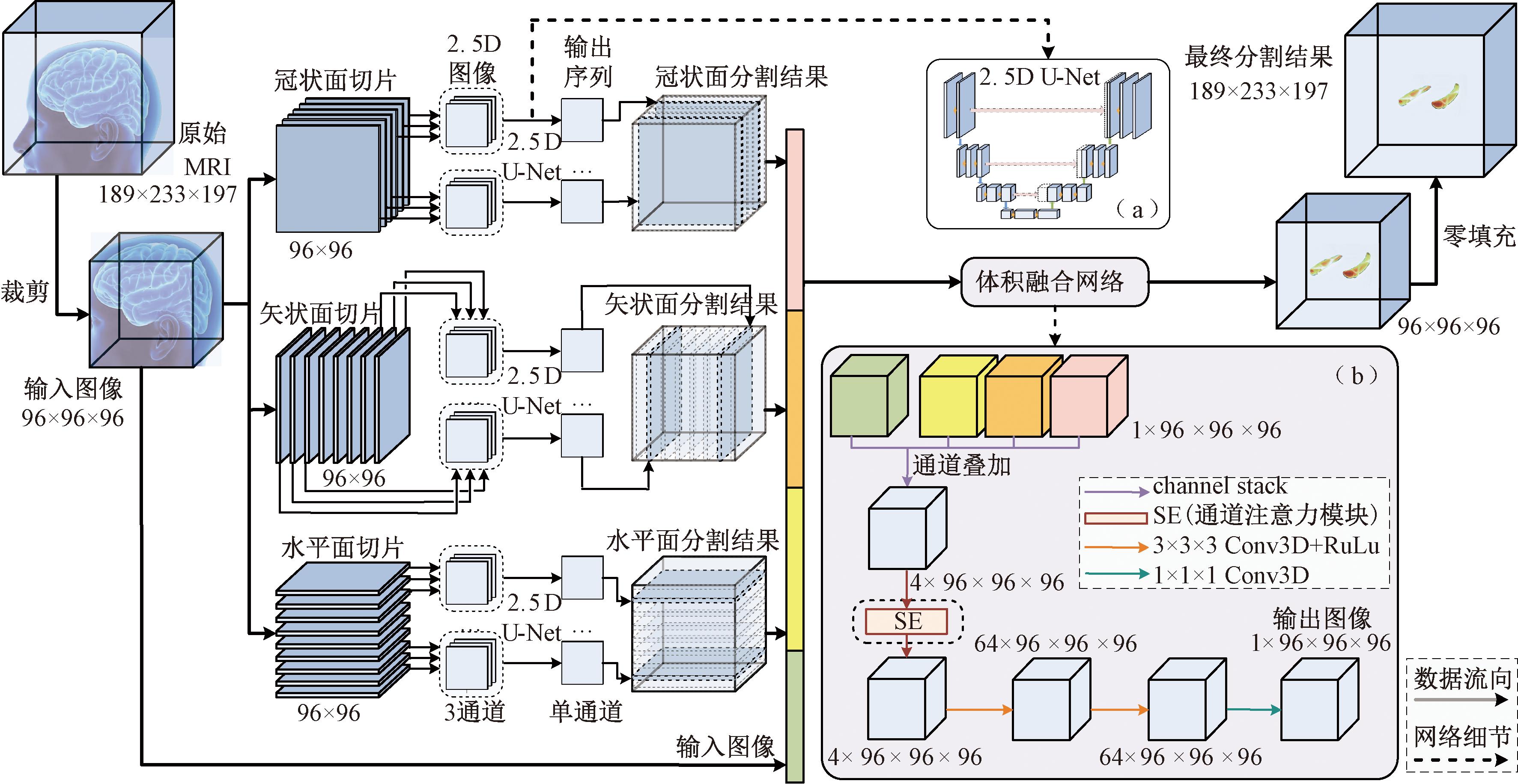
图2 MVF-2.5D U-Net的详细架构
Figure 2 Detailed architecture of MVF-2.5D U-Net
(1)对原始的MRI图像进行预处理和裁剪,得到尺寸为96×96×96的输入图像。
(2)将输入图像沿3个视图方向上切片得到3组尺寸为96×96的2D切片序列,随后,将每张切片与两张相邻切片堆叠为三通道的2.5D图像。
(3)将2.5D图像逐一输入该视图方向上改进的2.5D U-Net模型中得到对应的单通道输出序列,网络结构如图2中(a)所示,随后将输出序列按原顺序堆叠回到96×96×96的尺寸作为该方向上的分割结果。
(4)将3个分割结果与原始图像堆叠成一张4通道的输入图像,经过体积融合网络得到相同尺寸的输出图像,如图2中(b)所示。
(5)通过零填充将输出图像恢复到与原始图像相同的尺寸,得到最终的分割结果。
2.1 改进的2.5D U-Net
本文对传统的2D U-Net进行了改进,如图3所示,以2.5D图像作为网络的输入,并在传统U-Net中的跳跃连接处加入了Triplet Attention模块,该模块采用了三重注意力跨维度交互机制,通过3个分支分别捕捉通道与高度(C和H)、通道与宽度(C和W)以及空间维度(H和W)的注意力权重,以增加网络在特征提取过程中对关键区域的关注程度。另外,该网络将传统2D U-Net的池化次数减少至3次,如果采用4次池化,最深层图像尺寸为6×6,分辨率过低,容易丢失图像的细节信息。
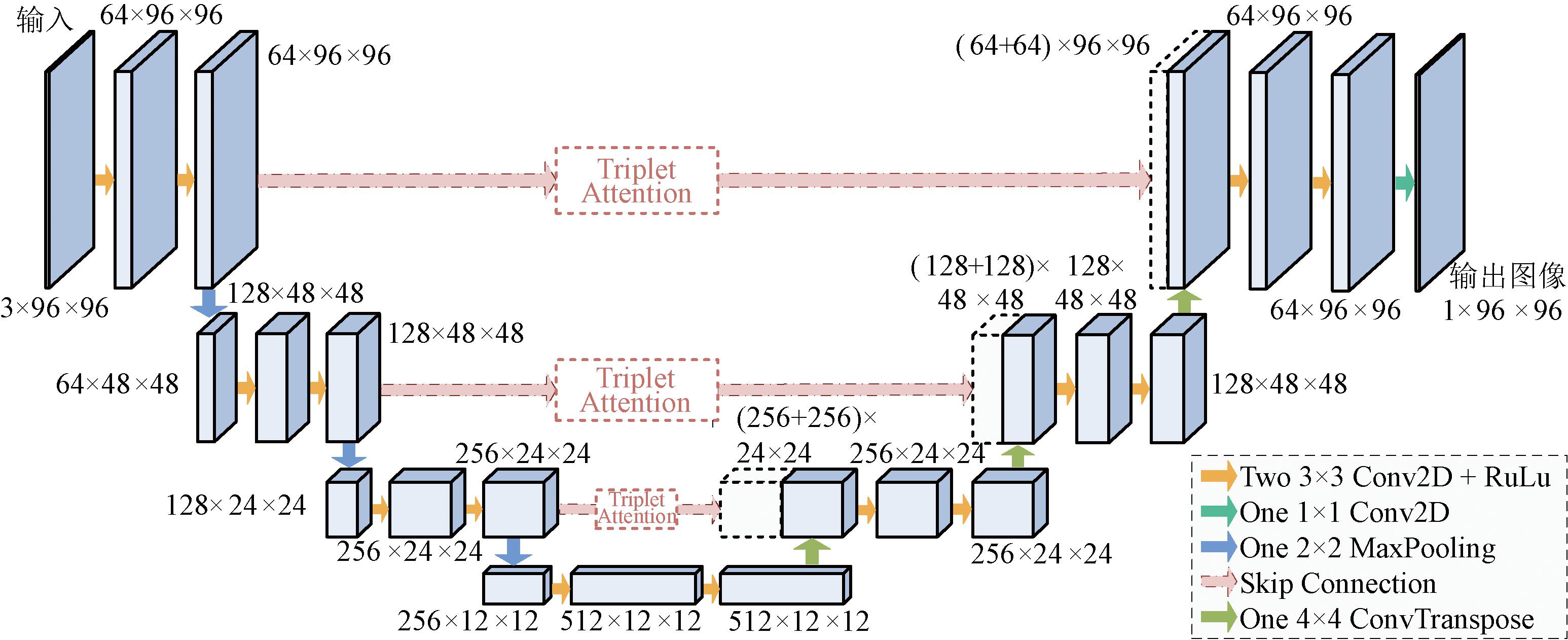
图3 改进的2.5D U-Net网络
Figure 3 Improved 2.5D U-Net network
2.2 多通道融合网络
第二阶段的体积融合网络将第一阶段输出的3个海马体分割图像进行融合,如图2中(b)所示。3D图像中同一体素在不同视图切片中的对比度和可供参考的信息有较大的差别。多网络融合策略可以充分利用不同视角包含的信息,还可以去除单个网络在分割时出现的外围噪点,消除随机误差。在此基础上,额外地引入原始图像作为融合网络输入的一部分,类似于引入残差连接的做法,这样能够有效地防止网络因深度过大而在处理过程中丢失有用的信息。该网络还在输入端引入了SE模块来自动学习不同通道的重要程度。SE模块是一种通道注意力模块,该模块通过训练,自动学习不同通道的注意力权重,以调节网络对不同通道的关注程度,实现网络对不同图像分割结果及原始图像重要度的分配。为了保证融合网络的非线性处理能力,在注意力模块后又增加了多个卷积运算单元,最终通过1×1卷积把特征图转化为单通道的分割图像。
2.3 通道扩充
将实验所使用的MRI处理成一系列的单通道灰度切片后,把其中一张切片与两张相邻切片堆叠,可以组成一张3通道的2.5D图像,图4展示了将某样本水平面第95,96,97张2D切片组合为3通道2.5D图像的过程。通过观察可发现,图4中左侧第95,96,97张切片中的脑组织在右侧合成后的2.5D图像中分别呈现出红、绿、蓝3种颜色,类似于3通道的彩色RGB图像,但此时3个通道不再表示颜色信息,而是空间信息。2.5D图像仍使用2D网络进行处理,这种通道扩充方式增加了样本中空间信息的利用率,在一定程度上弥补了2D分割网络无法利用空间信息的劣势,同时避免了3D网络沉重的计算负担。该方法仅对输入图像进行扩充,输出图像以及训练所用的标签仍是中心切片所对应的单通道的分割结果,相邻切片仅在输入网络时被作为参考。
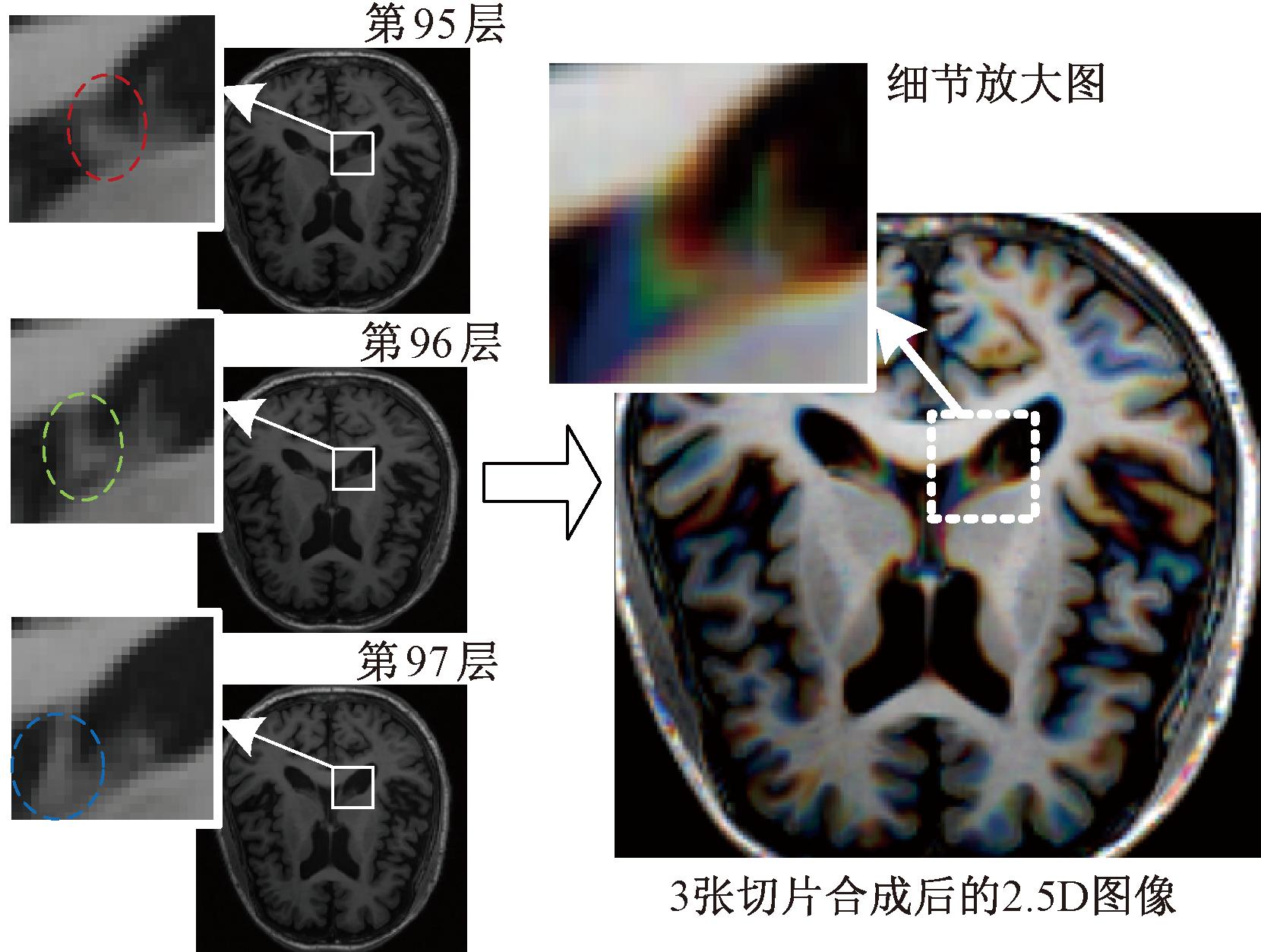
图4 2.5D图像示意图
Figure 4 Schematic diagram of 2.5D image
2.4 预处理
图5为数据集中海马体图像的位置。
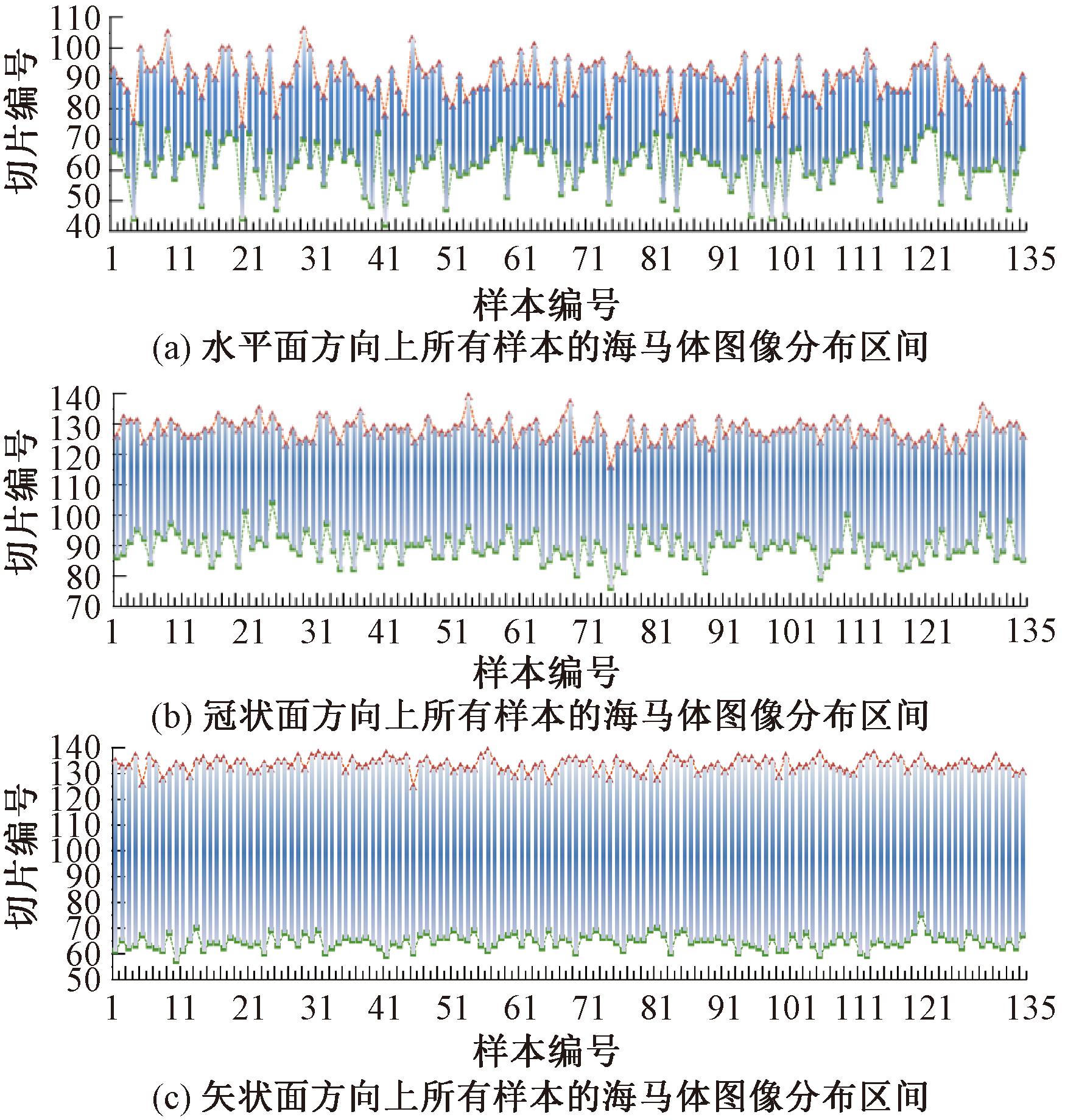
图5 数据集中海马体图像的位置
Figure 5 Location of the hippocampus image in the dataset
为了增强网络的性能,对MRI进行了归一化处理和裁剪,使其尺寸从189×233×197减小至96×96×96。通过分析135例数据中的样本标签,发现海马体图像在3个方向上的分布区间相对固定,如图5所示,横坐标表示135例样本的编号,纵坐标表示切片的编号。首先,分别在3个方向上根据每例样本海马体出现在该方向上的起点和终点,计算出该样本海马体所处区间的中点;其次,取135个样本中点的平均值,并将该值±48分别作为该方向上裁剪框的起点和终点;最后,将其与所有样本海马体在该方向上端点的最值进行比较,如表1所示。由表1可知,根据该方法所选的裁剪区间既包含所有样本的海马体,且裁剪框的边界距离最小值和最大值仍有一定裕度。
表1 裁剪框范围与HarP数据集中所有海马体图像端点最值的对比
Table 1 Comparison between the cropping box dimensions and the extremal values of hippocampal image endpoints in the HarP dataset
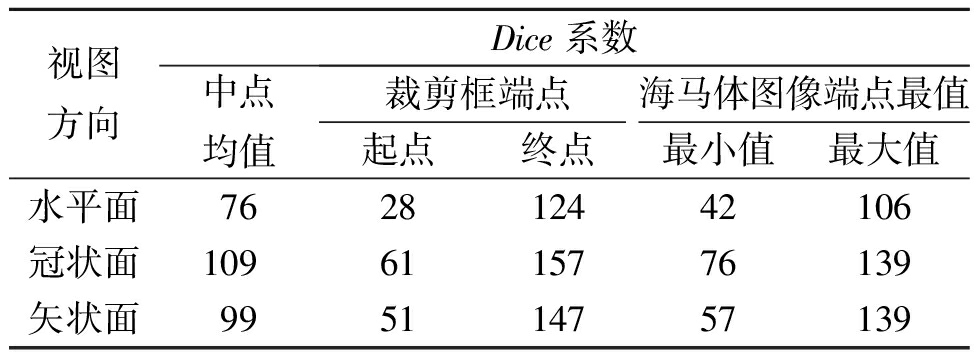
视图方向Dice系数中点均值裁剪框端点海马体图像端点最值起点终点最小值最大值水平面762812442106冠状面1096115776139矢状面995114757139
2.5 模型训练
在第一阶段网络训练时,由于矢状面、冠状面、水平面的切片具有较大差异,根据不同视图方向上的切片独立训练3个子网络,每个网络专注于一个视图方向上的分割任务。在第一阶段的网络训练完成后,固定第一阶段3个网络的参数。随后,将训练集和验证集中的图像和标签同时进行镜像和适当的位移处理,以扩充数据集。将扩充后的数据集输入3个训练好的第一阶段网络得到3组粗分割结果并将其作为第二阶段融合网络的输入,用于第二阶段的网络训练。经过实验验证,如果不进行扩充,而是将原训练集和验证集重复输入第一阶段网络,那么所得到的粗分割结果更优,不符合真实性,不利于充分挖掘融合网络的潜能。无论采用以上哪种训练方法,在模型训练过程中都没有接触测试集,只有在两阶段网络全部完成训练后,才会将测试集输入网络来评估网络的性能。
3 实验结果与分析
3.1 数据集
实验数据集采用阿尔茨海默病神经成像计划(Alzheimer′s disease neuroimaging initiative,ADNI)中EADC项目的HarP数据集[21],具体受试者信息如表2所示。通过5名专家对海马体图像进行手动分割标注,并逐层检查,在需要时进行校正,形成了金标准,MRI及标签的尺寸均为189×233×197。
表2 HarP数据集中135位受试者的信息
Table 2 Information from the 135 subjects in the HarP dataset

病程类别总数量男性数量女性数量年龄分布中位数平均数最小值最大值正常(HC)46222474.5576.1861.189.7轻度认知障碍(MCI)46271975.5574.7060.487.4阿尔茨海默病(AD)45212473.1074.4562.789.7
3.2 实验平台
本实验基于Windows10操作系统,显卡型号为NVIDIA RTX 2080、显存为8 G,采用的编程语言为Python 3.6,深度学习框架为Pytorch 1.10.2,采用Adam优化器,初始学习率为0.001,动量衰减因子为0.9,迭代过程采用早停机制避免过拟合,第一、二阶段网络训练中的batch size分别设置为20和4。
3.3 验证方式及评价指标
实验采用5折交叉验证的方法来评估模型的性能,采用Dice系数和豪斯多夫距离HD来衡量分割结果的准确率和稳定性。在该任务中,标签及预测结果通常由值为0或1的两种体素组成。Dice系数通常用来度量两个集合的相似性,而HD通常用来度量边界距离及稳定性。其计算公式分别为
(1)
(2)
(3)
HD(A,B)=max{h(A,B),h(B,A)}。
(4)
式中:A、B分别为标签及预测结果中海马体图像所在体素的集合;|A|、|B|分别为标签和预测结果中海马体图像所在区域的体素总数;|A∩B|为两部分重叠区域的体素总数;d(a,b)为a、b之间的欧氏距离。Dice系数越接近1,或HD越小,预测结果越准确。
实验采用BCEDiceLoss作为损失函数,该损失函数为DiceLoss(即1-Dice)与二元交叉熵损失函数BCELoss之和。BCELoss的计算公式为
BCELoss=-[ylog p+(1-y)log(1-p)]。
(5)
式中:p为分割结果中某一体素模型预测为1的概率;y为该体素在标签中对应的值,为二值变量0或1。其中,BCELoss被用于单独评估每个像素的损失,然后再对所有像素求平均值得到该样本的总损失。最终,BCEDiceLoss的计算公式为
BCEDiceLoss=DiceLoss+BCELoss。
(6)
3.4 实验结果
为了验证模型的性能,将本文方法与传统方法进行了对比,并将某样本在不同网络下的分割结果以(75,74,125)位置的3视图和整体的3D视图进行了展示,如图6所示,该位置的海马体从各方向来看边缘清晰,形态较为典型。由图6可见,3个经典的2D U-Net模型对海马体边界的分割不够清晰,且存在异常的噪点,如白色箭头所示,这些噪点从3D视图来看更为明显,如白色方框所示,同时由于缺少空间信息,仅通过单个切片来分割很容易在狭窄区域出现漏判,如蓝色箭头所示;3D U-Net模型从各方向来看基本无噪点,且边界较为光滑,但在一些狭窄区域上容易漏判,如红色箭头所示;而本文方法不仅边界清晰,且在狭窄区域上的分割效果也优于其他方法,更加接近专家手动分割结果。

图6 不同模型的分割结果
Figure 6 Segmentation results of different models
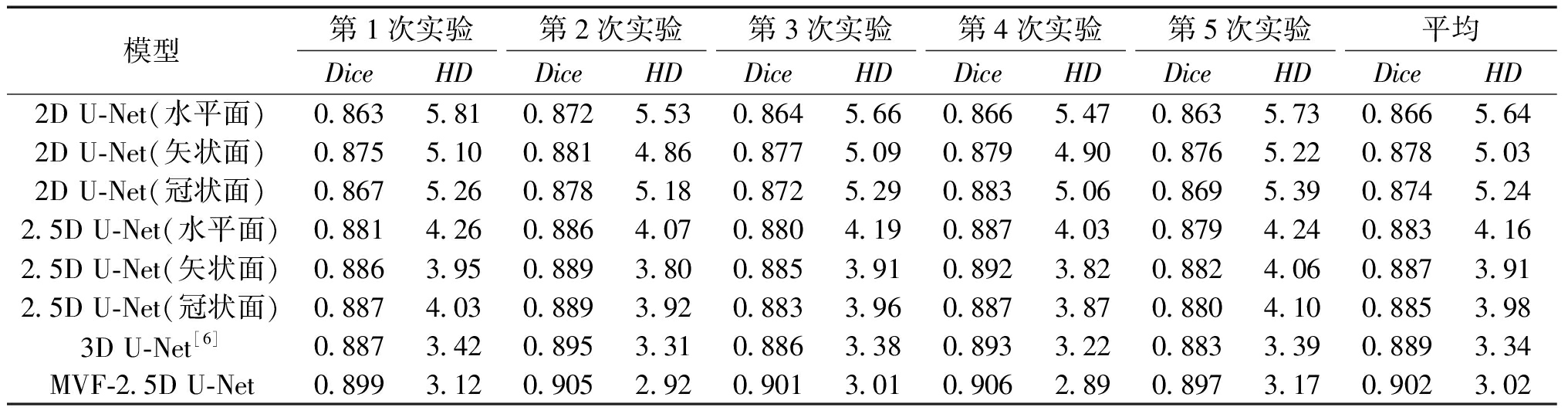
为了更好地验证各网络的性能,实验还加入了3个方向上的改进后的2.5D U-Net作为对照,不同模型的分割结果如表3所示。
表3 改进后的模型与几种原始U-Net模型在HarP数据集中的性能对比
Table 3 Performance comparison of the improved model and several original U-Net models in the HarP dataset
模型第1次实验第2次实验第3次实验第4次实验第5次实验平均DiceHDDiceHDDiceHDDiceHDDiceHDDiceHD2D U-Net(水平面)0.8635.810.8725.530.8645.660.8665.470.8635.730.8665.642D U-Net(矢状面)0.8755.100.8814.860.8775.090.8794.900.8765.220.8785.032D U-Net(冠状面)0.8675.260.8785.180.8725.290.8835.060.8695.390.8745.242.5D U-Net(水平面)0.8814.260.8864.070.8804.190.8874.030.8794.240.8834.162.5D U-Net(矢状面)0.8863.950.8893.800.8853.910.8923.820.8824.060.8873.912.5D U-Net(冠状面)0.8874.030.8893.920.8833.960.8873.870.8804.100.8853.983D U-Net[6]0.8873.420.8953.310.8863.380.8933.220.8833.390.8893.34MVF-2.5D U-Net0.8993.120.9052.920.9013.010.9062.890.8973.170.9023.02
由表3可知,本文提出的MVF-2.5D U-Net模型取得了最高的Dice系数和最低的豪斯多夫距离HD,其分割准确率与稳定性均优于所有对照方法。此外,改进的2.5D U-Net与原始的2D U-Net相比,在3个视图方向上的两项评价指标也均有改善,其中豪斯多夫距离HD的变化更为明显,这主要是由于2.5D U-Net通过引入相邻切片中的上下文信息使得一些不规则噪点大幅减少。
此外,还测试了模型参数所占的内存、运行所需的最低内存和分割一张完整的3D MRI所需的时间,如表4所示。在计算效率上,2D网络和本文方法需要处理多张切片,导致检测时间增加,这也是该方法的唯一缺点,但该任务并不需要过高的实时性,该缺点几乎可以忽略[9],并且通过GPU的并行处理可以加快检测过程,然而,2D模型的参数和所需内存都是最少的,且远小于3D模型[9]。3D模型提高了准确率,但是增加了约20倍的内存开销[22],其主要来自3D特征图在传播过程中占用的内存,不适合在资源受限的环境下应用。改进的2.5D U-Net通过引入轻量化的SE模块和2.5D输入,可在保持与2D U-Net几乎相同参数和内存的条件下提高分割准确率。MVF-2.5D U-Net在多视图融合时处理3D图像所需的内存虽然大于单个2.5D-UNet,但仍远小于3D U-Net所需的内存,且拥有更高的分割准确率,在资源受限的环境下,可先在其他计算机中训练,再部署或采用众数投票机制来替代融合网络,以进一步降低内存开销。以上实验证明本文提出的改进的2.5D U-Net和MVF-2.5D U-Net模型均具有较强的可行性。
表4 不同模型所需最低内存和检测时间对比
Table 4 Comparison of minimum memory required and detection time of different models
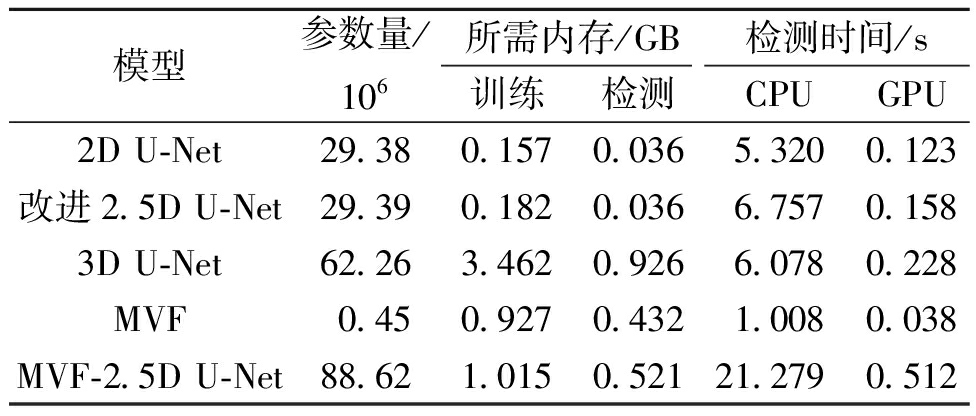
模型参数量/106所需内存/GB检测时间/s训练检测CPUGPU2D U-Net29.380.1570.0365.3200.123改进2.5D U-Net29.390.1820.0366.7570.1583D U-Net62.263.4620.9266.0780.228MVF0.450.9270.4321.0080.038MVF-2.5D U-Net88.621.0150.52121.2790.512
为了进一步验证本文提出的MVF-2.5D U-Net算法的优势,还将实验结果与最新的几种主流深度学习方法进行了对比,这些算法均在HarP数据集上进行实验,实验结果如表5所示。由表5可知,本文提出的MVF-2.5D U-Net模型与其他算法相比,在分割准确率上取得了更好的效果,可以更精确地实现大脑MRI的海马体图像分割任务。
表5 不同算法在HarP数据集中的性能对比
Table 5 Performance comparison of different algorithms in the HarP dataset

方法平均Dice系数Deep CNN EATL(2019)[23]0.901E2DHipseg(2021)[24]0.900DeepHarp(2021)[25]0.880RBS-Net(2023)[26]0.898HGM-cNet(2023)[27]0.895MVF-2.5D U-Net0.902
3.5 消融实验
为了验证体积融合网络VFN中各模块对网络性能的影响,本文设计了一组消融实验。其中,VFN-image移除了原始MRI图像,VFN-SE去除了通道注意力单元SE, VFN-conv删除了卷积运算单元。此外,实验还进一步验证了多层网络的效果,具体做法是在单层融合网络中的卷积运算单元中加入了池化层和反卷积层,以类比分割任务中常用的U型编码器-解码器架构,并分析层深对融合网络的影响,VFN-2、VFN-3、VFN-4分别对应2,3,4层卷积运算单元的变体,实验结果如表6所示。由表6可知,本文方法相比各对照组表现出了最好的性能,主要是因为原始MRI的加入能够弥补第一阶段网络分割时丢失的信息。通道注意力单元能在网络融合前自适应地学习不同通道所表示的先验信息的重要度权重,能够更好地利用粗分割结果。如果没有后续的卷积运算单元,网络的深度不够、对应的参数过少,其泛化能力受到了一定的约束,甚至会在训练时出现无法收敛的现象。但是,卷积运算单元并不是越复杂越好,随着网络深度的增加,网络性能变差,这是因为图像经过第一阶段的粗分割,已经能够得到较为准确的分割图像,如果融合网络的深度过大,不但会使分割图像在处理过程中出现较大的改变,还会导致其受到深层网络中低分辨率特征图的影响。
表6 不同体积融合网络变体在HarP数据集上的性能对比
Table 6 Performance comparison of different volume fusion network variants in the HarP dataset
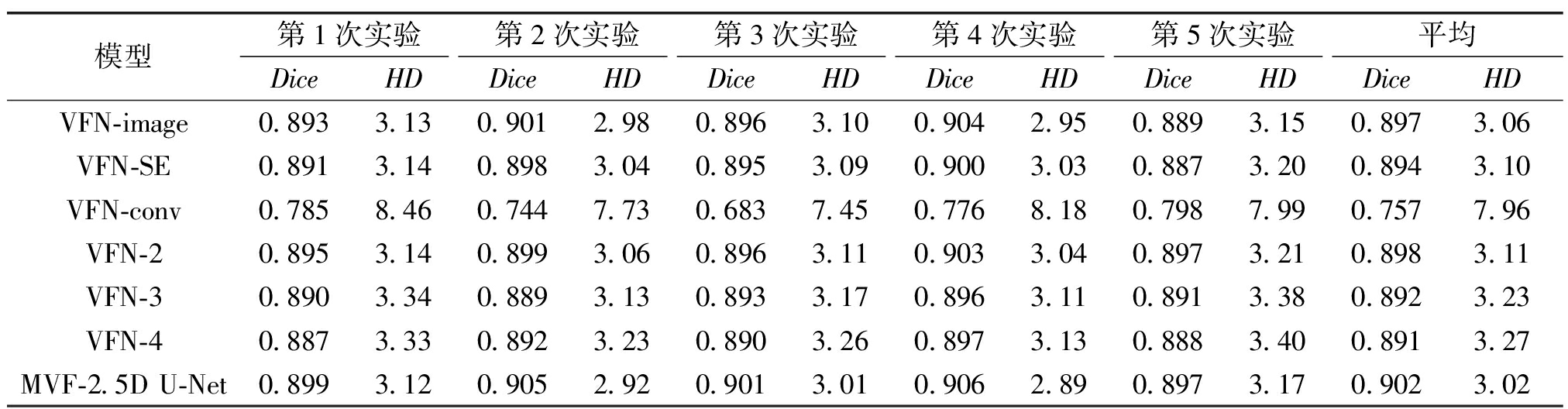
模型第1次实验第2次实验第3次实验第4次实验第5次实验平均DiceHDDiceHDDiceHDDiceHDDiceHDDiceHDVFN-image0.8933.130.9012.980.8963.100.9042.950.8893.150.8973.06VFN-SE0.8913.140.8983.040.8953.090.9003.030.8873.200.8943.10VFN-conv0.7858.460.7447.730.6837.450.7768.180.7987.990.7577.96VFN-20.8953.140.8993.060.8963.110.9033.040.8973.210.8983.11VFN-30.8903.340.8893.130.8933.170.8963.110.8913.380.8923.23VFN-40.8873.330.8923.230.8903.260.8973.130.8883.400.8913.27MVF-2.5D U-Net0.8993.120.9052.920.9013.010.9062.890.8973.170.9023.02
4 结论
本研究以大脑MRI海马体图像分割任务为背景,提出了一种基于CNN的分割+融合二段式海马体图像分割方法。该研究通过在公开数据集HarP上的多次交叉验证,取得了0.902的平均Dice系数和3.02的平均豪斯多夫距离,与其他方法相比有明显的提高,表明本文提出的海马体图像分割模型能够精准地实现海马体图像分割;同时,该方法相比于3D模型占用的内存较小,适用于内存受限的环境中。该方法通过自动分割替代传统的手动分割,只需对分割结果进行微调,极大地降低了医师的工作量,分割后的海马体图像可结合其他特征进一步用于阿尔茨海默病、癫痫等疾病的诊断。下一步工作还将在其他数据集(如HCUnicamp[24]、OASIS[25])上进行测试,以进一步验证该方法的鲁棒性及泛化能力。
[1] S MANN P G, IGLESIAS J E, GUTMAN B, et al. FreeSurfer-based segmentation of hippocampal subfields: a review of methods and applications, with a novel quality control procedure for ENIGMA studies and other collaborative efforts[J]. Human Brain Mapping, 2022, 43(1): 207-233.
MANN P G, IGLESIAS J E, GUTMAN B, et al. FreeSurfer-based segmentation of hippocampal subfields: a review of methods and applications, with a novel quality control procedure for ENIGMA studies and other collaborative efforts[J]. Human Brain Mapping, 2022, 43(1): 207-233.
[2] JOULES R, WOLZ R. Fully automatic deep-learning based segmentation of hippocampal subfields from standard resolution T1W MRI in Alzheimer′s disease[J]. Alzheimer′s &Dementia, 2023, 19: e076575.
[3] SACKL M, TINAUER C, URSCHLER M, et al. Fully automated hippocampus segmentation using T2-informed deep convolutional neural networks[J]. NeuroImage, 2024, 298: 120767.
[4] NI T W, XIE L X, ZHENG H J, et al. Elastic boundary projection for 3D medical image segmentation[C]∥Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition. Piscataway: IEEE, 2019: 2109-2118.
[5] LIU M H, LI F, YAN H, et al. A multi-model deep convolutional neural network for automatic hippocampus segmentation and classification in Alzheimer′s disease[J]. NeuroImage, 2020, 208: 116459.
[6] ÇIÇEK Ö, ABDULKADIR A, LIENKAMP S S, et al. 3D U-Net: learning dense volumetric segmentation from sparse annotation[C]∥Medical Image Computing and Computer-Assisted Intervention. Cham:Springer, 2016: 424-432.
[7] MANJON J V, ROMERO J E, COUPE P. A novel deep learning based hippocampus subfield segmentation method[J]. Scientific Reports, 2022, 12(1): 1333.
[8] KNUTH F, ADDE I A, HUYNH B N, et al. MRI-based automatic segmentation of rectal cancer using 2D U-Net on two independent cohorts[J]. Acta Oncologica, 2022, 61(2): 255-263.
[9] ZETTLER N, MASTMEYER A. Comparison of 2D vs. 3D U-Net organ segmentation in abdominal 3D CT images[EB/OL].(2021-07-08)[2024-07-16].https:∥doi.org/10.48550/arXiv.2107.04062.
[10] ROTH H R, LU L, SEFF A, et al. A new 2.5D representation for lymph node detection using random sets of deep convolutional neural network observations[C]∥Medical Image Computing and Computer Assisted Intervention. Cham: Springer International Publishing, 2014: 520-527.
[11] ROTH H R, LU L, FARAG A, et al. DeepOrgan: multi-level deep convolutional networks for automated pancreas segmentation[C]∥Medical Image Computing and Computer Assisted Intervention. Cham: Springer, 2015: 556-564.
[12] YU Q H, XIE L X, WANG Y, et al. Recurrent saliency transformation network: incorporating multi-stage visual cues for small organ segmentation[C]∥2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Piscataway:IEEE, 2018: 8280-8289.
[13] CHEN J, LU Y, YU Q, et al. TransUNet: Transformers make strong encoders for medical image segmentation[EB/OL].(2021-02-08)[2024-07-16].https:∥doi.org/10.48550/arXiv.2102.04306.
[14] 欧宇轩, 高敏, 赵地, 等. SA-TF-UNet: 基于空间注意力机制和Transformer的MRI海马体分割[J]. 中国图象图形学报, 2023, 28(10): 3191-3202.
OU Y X, GAO M, ZHAO D, et al. SA-TF-UNet: a Transformer and spatial attention mechanisms based hippocampus segmentation network[J]. Journal of Image and Graphics, 2023, 28(10): 3191-3202.
[15] OKTAY O, SCHLEMPER J, LE FOLGOC L, et al. Attention U-Net: learning where to look for the pancreas[EB/OL].(2018-04-18) [2024-07-16] .https:∥doi.org/10.48550/arXiv.1804.03999.
[16] WANG H, LEI C, ZHAO D, et al. DeepHipp: accurate segmentation of hippocampus using 3D dense-block based on attention mechanism[J]. BMC Medical Imaging, 2023, 23(1): 158.
[17] 魏明军, 王镆涵, 刘亚志, 等. 基于特征融合和混合注意力的小目标检测[J]. 郑州大学学报(工学版), 2024, 45(3): 72-79.
WEI M J, WANG M H, LIU Y Z, et al. Small object detection based on feature fusion and mixed attention[J]. Journal of Zhengzhou University (Engineering Science), 2024, 45(3): 72-79.
[18] 高宇飞, 马自行, 徐静, 等. 基于卷积和可变形注意力的脑胶质瘤图像分割[J]. 郑州大学学报(工学版), 2024, 45(2): 27-32.
GAO Y F, MA Z X, XU J, et al. Brain glioma image segmentation based on convolution and deformable attention[J]. Journal of Zhengzhou University (Engineering Science), 2024, 45(2): 27-32.
[19] ZHOU Y Y, XIE L X, SHEN W, et al. A fixed-point model for pancreas segmentation in abdominal CT scans[C]∥Medical Image Computing and Computer Assisted Intervention. Cham: Springer, 2017: 693-701.
[20] XIA Y D, XIE L X, LIU F Z, et al. Bridging the gap between 2D and 3D organ segmentation with Volumetric Fusion Net[C]∥Medical Image Computing and Computer Assisted Intervention. Cham: Springer, 2018: 445-453.
[21] BOCCARDI M, BOCCHETTA M, MORENCY F C, et al. Training labels for hippocampal segmentation based on the EADC-ADNI harmonized hippocampal protocol[J]. Alzheimer′s &Dementia,2015, 11(2): 175-183.
[22] AVESTA A, HOSSAIN S, LIN M D, et al. Comparing 3D, 2.5D, and 2D approaches to brain image auto-segmentation[J]. Bioengineering, 2023, 10(2): 181.
[23] ATALOGLOU D, DIMOU A, ZARPALAS D, et al. Fast and precise hippocampus segmentation through deep convolutional neural network ensembles and transfer learning[J]. Neuroinformatics, 2019, 17(4): 563-582.
[24] CARMO D, SILVA B, INITIATIVE A D N, et al. Hippocampus segmentation on epilepsy and Alzheimer′s disease studies with multiple convolutional neural networks[J]. Heliyon, 2021, 7(2): e06226.
[25] NOBAKHT S, SCHAEFFER M, FORKERT N D, et al. Combined atlas and convolutional neural network-based segmentation of the hippocampus from MRI according to the ADNI harmonized protocol[J]. Sensors, 2021, 21(7):2427.
[26] CHEN Y, YUE H L, KUANG H L, et al. RBS-Net: hippocampus segmentation using multi-layer feature learning with the region, boundary and structure loss[J]. Computers in Biology and Medicine, 2023, 160: 106953.
[27] ZHENG Q, LIU B, GAO Y, et al. HGM-cNet: integrating hippocampal gray matter probability map into a cascaded deep learning framework improves hippocampus segmentation[J]. European Journal of Radiology, 2023, 162: 110771.