注意缺陷多动障碍(attention deficit hyperactivity disorder,ADHD)是一种常见的儿童精神疾病,主要表现为注意力不集中、多动等。目前,ADHD的诊断主要依靠专业医生根据临床经验和一些行为量表进行。这种方式具有较强的主观性,并且由于ADHD病理机制复杂[1],可能会增加误诊的风险,因此需要借助客观的辅助诊断技术。近年来,深度学习在疾病辅助诊断领域得到广泛应用[2]。深度学习具有强大的信息挖掘能力,不仅可以得到较好的ADHD疾病分类结果,还能完成对ADHD相关的生物特征学习与生物标识的检测。
目前,用于进行ADHD辅助诊断研究的数据大致包括脑电图生物信号(electroencephalogram,EEG)和磁共振成像。一些基于EEG的ADHD分类研究取得了较好的分类表现[3],但其不能揭示和ADHD患病相关的大脑异常部位。相比之下,使用磁共振成像技术得到的静息态功能磁共振图像(resting-state functional magnetic resonance imaging,rs-fMRI)对大脑中的异常活动具有良好的定位能力。因此,rs-fMRI成为一种研究人员广泛使用的数据。其中,基于rs-fMRI体素的ADHD分类研究准确率不高[4],但rs-fMRI的衍生数据,例如低频波动振幅(amplitude of low-frequency fluctuation,ALFF)、区域一致性和功能连接(functional connectivity,FC)等,展现出较好的分类性能。基于FC的研究甚至已经达到99.8%的高准确率[5],基于ALFF的ADHD分类研究目前只获得75%的准确率[6]。但是,ALFF是直接描述大脑某一区域活动强度的数据,相对于FC描述大脑区域之间的同步性更客观实际。因此,本文使用ALFF进行ADHD分类研究。
在ADHD的分类研究中,基于机器学习方法,例如支持向量机递归特征消除[7](support vector machine with recursive feature elimination,SVM-RFE)方法,能从输入数据中提取典型特征。但是,这类基于机器学习的ADHD分类算法并不能有效地拟合输入特征和ADHD疾病的关系,分类性能往往较差。与之相比,深度学习模型能够自动适应不同任务和场景,还能结合注意力策略来优化模型性能。例如,Riaz等[8]提出使用神经网络模拟ADHD的功能连接性的方法,但性能受噪声影响较大。Liu等[9]提出结合去噪自编码器和卷积神经网络的方法,用于提取rs-fMRI的空间特征,忽略了数据中的时间信息。Qiang等[10]在提取rs-fMRI数据中空间特征的基础上,使用门控循环单元提取时间特征,存在特征拟合不足的问题。Qiang等[11]提出时空注意力机制,并应用于时空特征提取网络,但是在寻找生物标识方面有局限性。目前,基于二分假设的ADHD研究方法如Att-AENet[12],已经达到极高的准确率,并且具有生物标识检测的能力。但是,采用机器学习和深度学习相结合的方法,特征提取数目需人为设置,不能实现ADHD特征的端到端学习,这降低了模型学习效率,还可能导致模型性能下降,并且模型无法一体化[13]。因此,本文提出基于二分假设端到端深度学习ADHD分类模型,旨在探索ALFF数据对ADHD疾病特征的表达能力,寻找边缘系统上的ADHD生物标识,并克服输入特征数目需人为设置的问题。
本文的主要贡献如下:构建带有注意力的二分假设端到端深度学习分类模型Deep-AENet;在二分假设框架下,使用注意力模块实现特征加权功能,形成端到端的特征学习;以边缘系统的ALFF数据作为输入特征,完成生物标识检测任务。
1 相关研究
1.1 边缘系统对ADHD疾病的影响
边缘系统是影响记忆和学习的大脑中心集合,在调节情绪和情感方面起着重要作用[14]。它包含许多重要的脑区,如海马体、丘脑、尾状核和前扣带回等,在以往的研究中这些脑区的异常与ADHD相关。例如,Ugarte等[15]在神经解剖研究中发现ADHD患者的海马体皮下质减少。海马体是短时记忆的存储转换和定向主要功能区,因此海马体异常可能会对ADHD患者造成注意力不集中等影响。此外,Bonath等[14]发现患有ADHD儿童的前扣带回减少,则表明ADHD与前扣带回相关。因此,边缘系统和ADHD疾病之间存在密切关系,进行基于边缘系统的ADHD分类研究具有重要意义。
1.2 二分假设框架
二分假设是一种基于假设检验的方法,能做出两种互斥的假设,从而影响网络模型对于输入数据的特征学习,是数据分析中的有效策略。基于二分假设的方法被广泛应用于ADHD的分类研究。例如,Tang等[16]在二分假设框架下提出一种子空间聚类方法,获得约90%的ADHD分类准确率。另外,基于l2范数的线性判别分析方法[17]在二分假设框架下进行ADHD特征学习,提高了鲁棒性,有效减轻噪声对ADHD分类的干扰。AENet网络[5]使用SVM-RFE进行典型特征选择,在ADHD-200各数据库上的准确率均超过99.3%。Chen等[12]提出的Att-AENet模型能在二分假设框架下使用注意力完成检测生物标识的任务。这些基于二分假设的方法很好地解决了rs-fMRI数据的噪声干扰和数据不足问题。因此,在二分假设框架的基础上进行ADHD分类是十分重要的。
2 本文方法
2.1 二分假设端到端ADHD分类算法
所提出的二分假设端到端ADHD分类模型如图1所示。在对测试数据做出H0和H1假设后(H0是健康对照假设,H1是ADHD假设),送入后续网络。该模型包括两个步骤,即特征提取和ADHD决策。
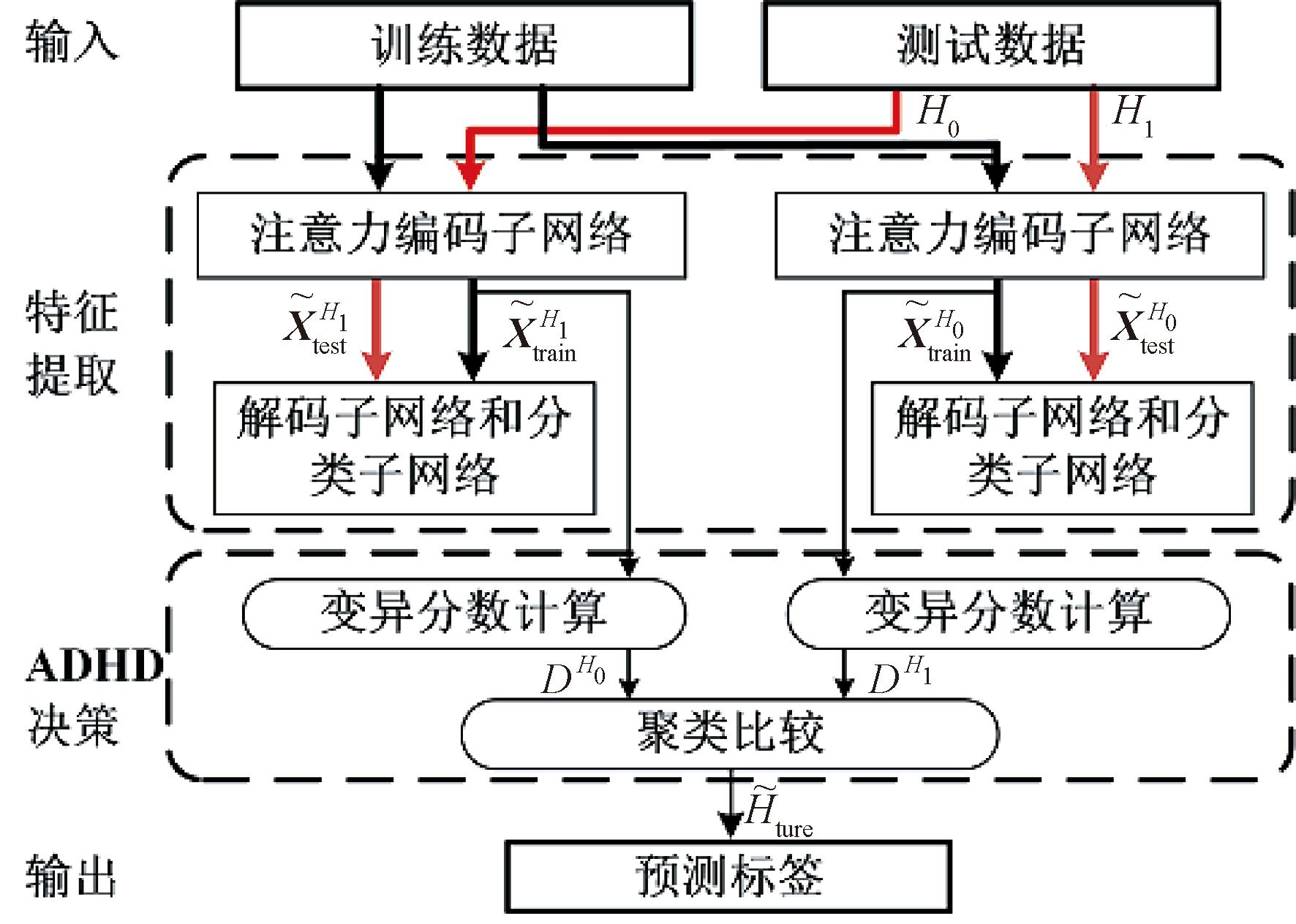
图1 基于二分假设端到端深度学习的ADHD分类模型
Figure 1 ADHD classification model based on binary hypothesis end-to-end deep learning
特征提取用以实现端到端的特征学习。注意力编码子网络是一个特征提取器,用来提取判别特征,得到不同假设下训练数据的特征![]() 和
和![]() 和测试数据的特征
和测试数据的特征![]() 和
和![]() 其中,注意力模块通过自适应化学习,强化分类贡献度高的特征权重。分类子网络用来评价特征学习的效果。
其中,注意力模块通过自适应化学习,强化分类贡献度高的特征权重。分类子网络用来评价特征学习的效果。
ADHD决策用来进行测试数据的标签预测。接受不同假设下的训练数据特征![]() 和
和![]() 对两组特征分别进行变异分数(DH0和DH1)计算,然后比较不同假设(H0和H1)的聚类性能,其中变异分数较小假设对应的假设标签即预测标签
对两组特征分别进行变异分数(DH0和DH1)计算,然后比较不同假设(H0和H1)的聚类性能,其中变异分数较小假设对应的假设标签即预测标签![]()
2.2 特征提取
特征提取网络结构由注意力编码子网络、解码子网络和分类子网络构成,如图2所示。ALFF数据送入网络之后,首先通过注意力模块来获得权重,随后加权ALFF被用于特征提取。提取到的特征被送入解码子网络和分类子网络,送入解码子网络的特征用于ALFF数据的重建,产生重建损失对所提取的特征进行优化;送入分类子网络的特征用于测试数据的标签预测,产生预测损失对所提取的特征进行优化。特征提取阶段由三重损失函数联合进行监督优化。经过三重优化后,训练数据的特征![]() 和
和![]() 被送入ADHD决策。
被送入ADHD决策。
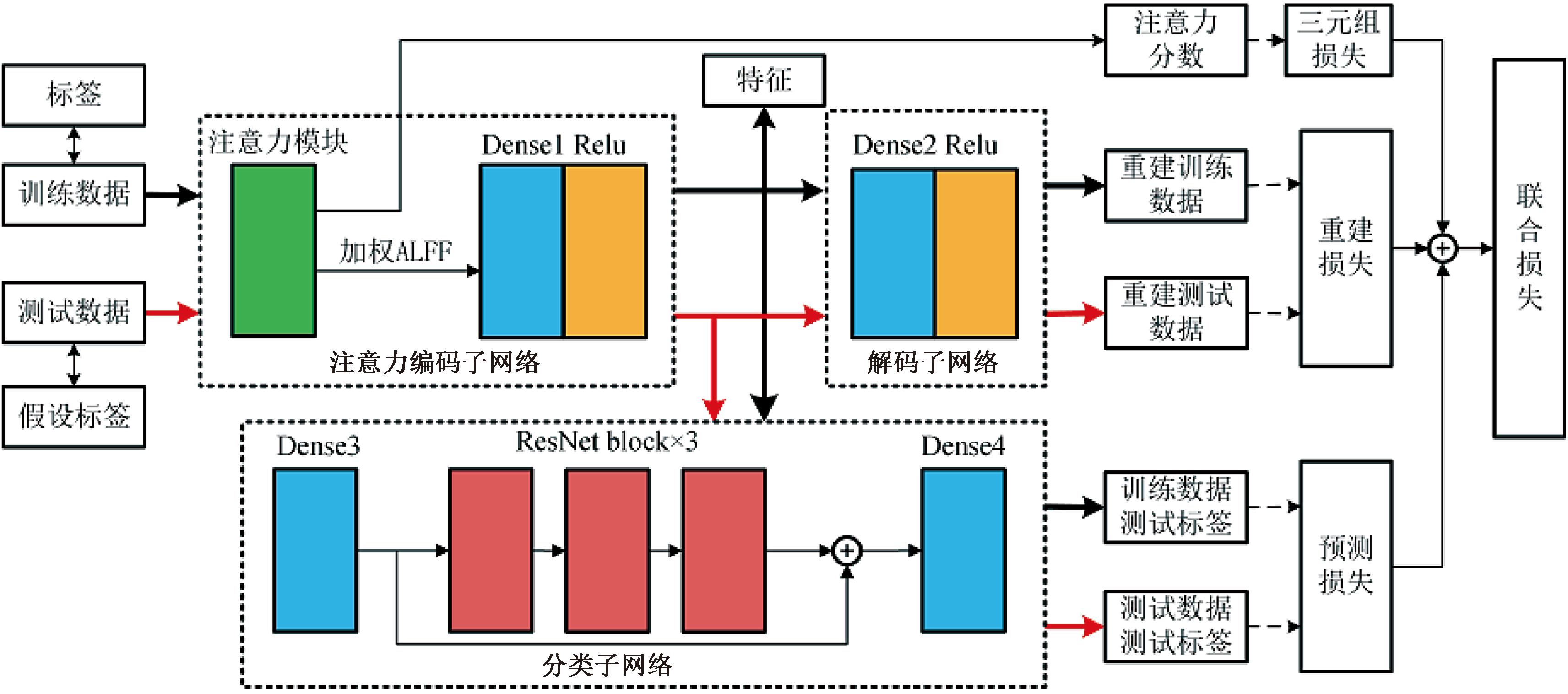
图2 Deep-AENet的特征提取网络结构
Figure 2 Feature extraction network structure of Deep-AENet
在注意力编码子网络设置了一个注意力模块。该注意力模块不仅能使网络重点关注ALFF中分类贡献高的数据,而且有助于完成后续ALFF数据的ADHD生物标识检测任务。该注意力模块结构如图3所示,由全连接层(Dense)、Relu激活层和Softmax激活层组成。其输入是24×N的ALFF数据,经过训练后产生24×N的注意力分数,并作用于ALFF数据,最后经过批处理归一化(BN)层,形成加权ALFF数据作为注意力模块的最终输出。
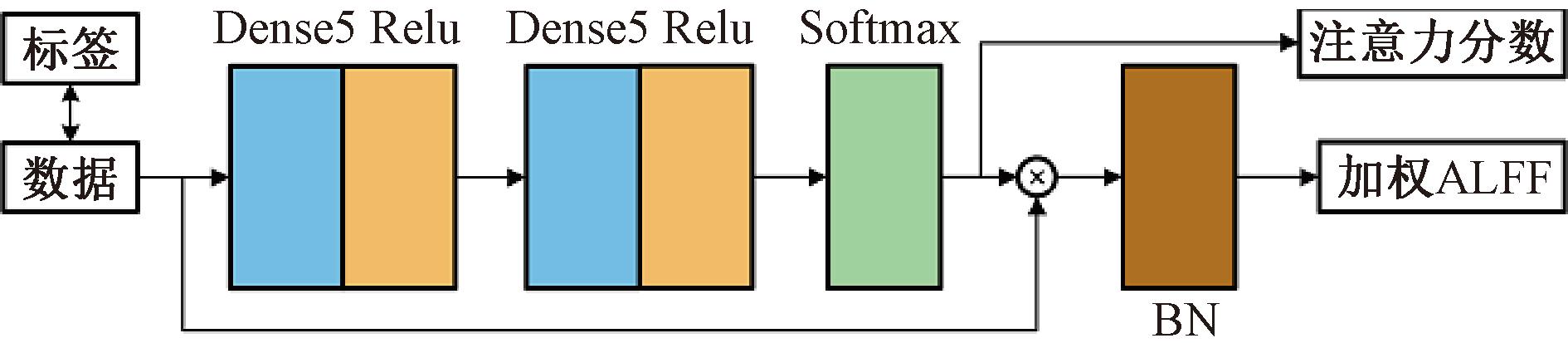
图3 注意力模块结构图
Figure 3 Attention block structure
网络中的全连接层参数如表1所示。此外,因为输入ALFF数据是正数,Deep-AENet在解码子网络有一个额外的Relu激活层,目的是保证重建ALFF的非负性。分类子网络的其他网络配置可以在文献[5]中找到。
表1 Deep-AENet的全连接层参数设置
Table 1 Fully connected layer parameters of Deep-AENet
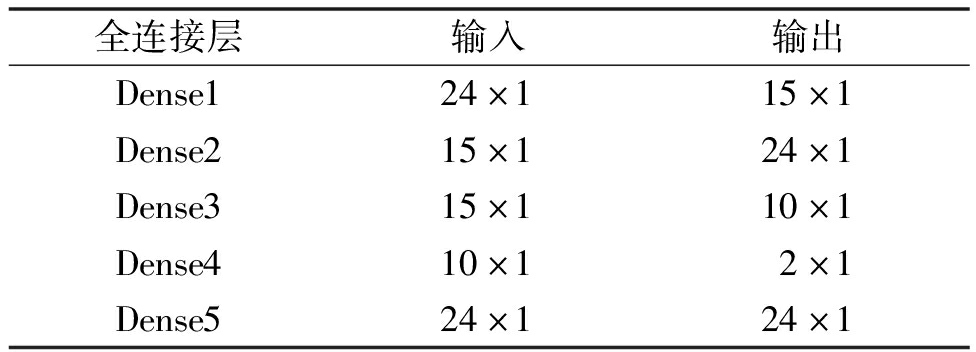
全连接层输入输出Dense124×115×1Dense215×124×1Dense315×110×1Dense410×12×1Dense524×124×1
2.3 ADHD决策
ADHD决策模块的任务是做出测试数据的预测标签![]() 通过对从注意力编码子网络获得的训练数据特征
通过对从注意力编码子网络获得的训练数据特征![]() 和
和![]() 进行计算,评估其对应假设的聚类性能。在某一假设下(H0和H1),特征
进行计算,评估其对应假设的聚类性能。在某一假设下(H0和H1),特征![]() 按照标签划分为两个子集,即ADHD患者子集
按照标签划分为两个子集,即ADHD患者子集![]() 和健康对照组子集
和健康对照组子集![]() train=
train=![]() ,其中na和nc分别为ADHD患者人数和健康对照组人数,每个子集的类内距离Dintra和类间距离Dinter由式(1)计算:
,其中na和nc分别为ADHD患者人数和健康对照组人数,每个子集的类内距离Dintra和类间距离Dinter由式(1)计算:
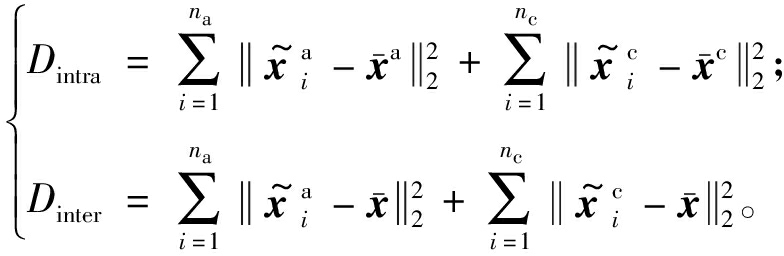
(1)
式中:![]() 和
和![]() 分别为
分别为![]() 和
和![]() 的平均特征。训练数据在不同假设下的变异分数DH0或DH1为
的平均特征。训练数据在不同假设下的变异分数DH0或DH1为
(2)
在错误假设下,具有错误标签的测试数据是一种噪声数据。在特征提取阶段会干扰网络的特征学习,相应的特征不能被适当地聚类,会导致变异分数增大。因此,具有较小变异分数对应的假设标签即为测试数据的预测标签:
(3)
式中:DH0为H0假设下的变异分数;DH1为H1假设下的变异分数。
2.4 网络设置
注意力模块用于对输入ALFF进行加权。使用三元组损失函数度量ADHD患者特征和健康对照组特征的注意力差异:
(4)
式中:N=na+nc为子数据库受试者数目;[·]+为取正操作;α为一个大于零的常数;![]() 为作为锚定样本的第i个受试者的注意力分数,其余N-1个样本根据标签分为两组,分别是与
为作为锚定样本的第i个受试者的注意力分数,其余N-1个样本根据标签分为两组,分别是与![]() 标签相同的正样本组
标签相同的正样本组![]() 和与
和与![]() 标签相反的负样本组
标签相反的负样本组![]()
在注意力编码子网络和解码子网络中采用均方误差损失来优化ALFF与其重建物之间的误差:
(5)
式中:X为输入![]() 为ALFF的重建物。
为ALFF的重建物。
分类子网络使用均方误差损失衡量模型的预测性能:
(6)
式中:yi为训练样本真实标签或测试数据的假设标签;![]() 为样本的预测标签。
为样本的预测标签。
最后在整个网络训练过程中由上述3个损失函数联合组成L监督:
L=Lt+Lr+Lp。
(7)
式中:Lt为三元组损失;Lr为重建损失;Lp为预测损失。
3 实验与分析
3.1 数据和预处理
实验数据来自ADHD-200数据库[18],分别为New York University (NYU)子数据库、Kennedy Krieger Institute(KKI)子数据库、Peking University(PU)子数据库和NeuroIMAGE(NI)子数据库。这些子数据库的样本信息如表2所示。各数据库超参数设置如表3所示,所有子数据库优化器均使用Adam优化器。
表2 ADHD-200样本信息表
Table 2 Sample information of ADHD-200
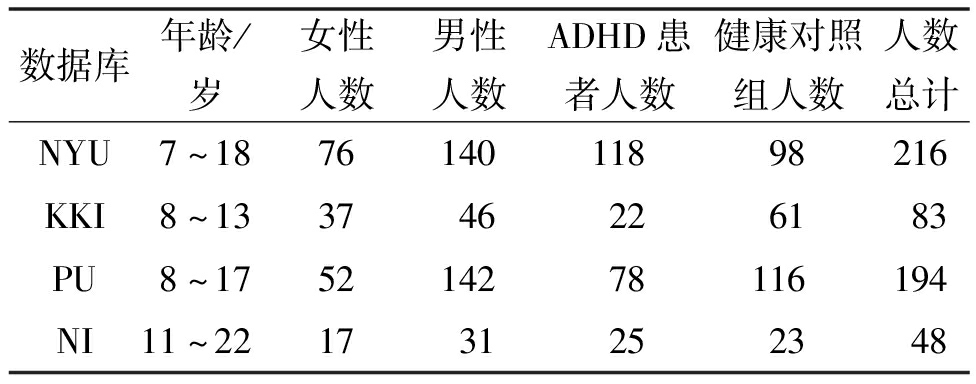
数据库年龄/岁女性人数男性人数ADHD患者人数健康对照组人数人数总计NYU7~187614011898216KKI8~133746226183PU8~175214278116194NI11~221731252348
表3 Deep-AENet模型超参数设置
Table 3 Experimental hyperparameters of Deep-AENet
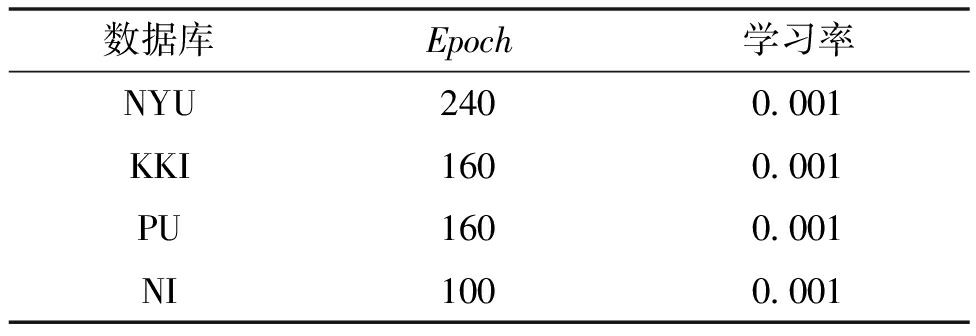
数据库Epoch学习率NYU2400.001KKI1600.001PU1600.001NI1000.001
数据处理包括预处理步骤,以及将血氧水平依赖(blood oxygen level dependent,BOLD)信号转为ALFF数据。集成的预处理工具[19]中进行的步骤包括:去除rs-fMRI数据的前4个时间点、切片时间校正、运动校正、体素配准、滤波和平滑。预处理之后获得BOLD信号,然后使用傅里叶变换将BOLD信号转换到频域,并计算低频波动的功率[20]。采用自动解剖标记图谱[21](AAL116)对大脑进行分组获得90个大脑区域作为感兴趣区域。最后在感兴趣区域上取平均值来获得脑区ALFF值,得到90×N的ALFF数据。为了探索边缘系统对ADHD分类的影响,只使用与边缘系统相关的24个脑区的24×N数据进行实验。
在ADHD-200数据库上的NYU、KKI、PU和NI、PU子数据库验证本文的方法。在验证过程中,所有子数据库均使用留一交叉验证方法评估ADHD分类准确性,该法用于评估小数据库模型十分有效。为了消除偶然因素和网络参数带来的影响,在进行20次重复实验后统计相关指标的平均结果。Deep-AENet的实验结果如表4所示,其中AUC为曲线下面积。
表4 Deep-AENet的分类表现
Table 4 Classification performance of Deep-AENet
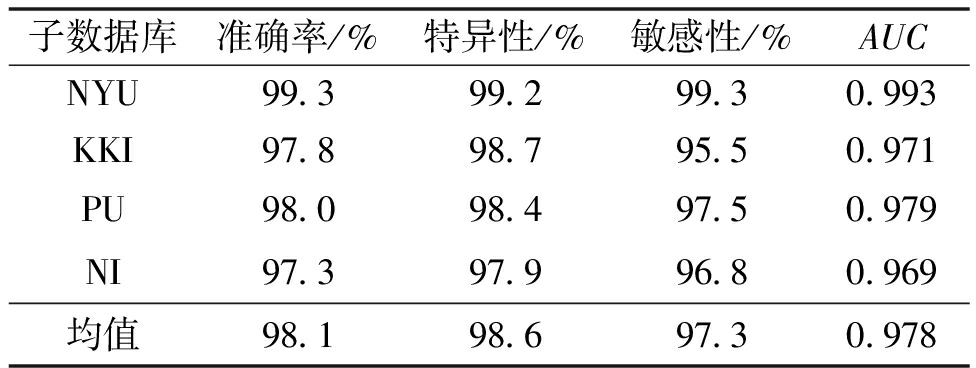
子数据库准确率/%特异性/%敏感性/%AUCNYU99.399.299.30.993KKI97.898.795.50.971PU98.098.497.50.979NI97.397.996.80.969均值98.198.697.30.978
由表4可知,Deep-AENet在各数据库上均取得较好的结果,ADHD分类准确率均在97.3%以上。在NYU子数据库上比在其他3个子数据库上获得更好的表现,这是因为在深度学习中,模型的分类表现一定程度上与样本量成正比。
此外,由于KKI数据库数据分布很不平衡(ADHD患者与健康对照组人数之比为22∶61),这不仅会影响分类表现,还会导致特异性与敏感性绝对差值变大。从平均AUC为0.978来看,Deep-AENet模型ADHD分类性能优秀。
3.2 方法的比较
本文比较了ADHD分类中基于深度学习的方法和基于二分假设框架的方法,如表5所示。由表5可知,在这些方法中,AENet和Att-AENet实现了最佳的平均准确率,并找到了ADHD相关的生物标识。从结构上看,不同于监督分类的方法,二分假设框架是一个半监督模型,能有效避免测试数据的有效特征在训练数据未被完全表达的情况,而这会影响ADHD分类表现。因此,本文采用了这一优秀的结构。从所使用的特征数目来看,AENet和Att-AENet方法中使用的特征数目为50,而所提出的Deep-AENet中使用的特征数目仅为24,在使用特征数不足其他二分假设模型一半的情况下,Deep-AENet取得了近似的结果,这表明本文方法对ADHD疾病特征进行了有效学习。从网络架构来看,本文方法具有端到端的结构,网络实现了特征从输入到输出的自动学习和优化,简化了学习过程,提高了学习效率。
表5 Deep-AENet与其他模型的性能比较
Table 5 Performance comparison between Deep-AENet and other models
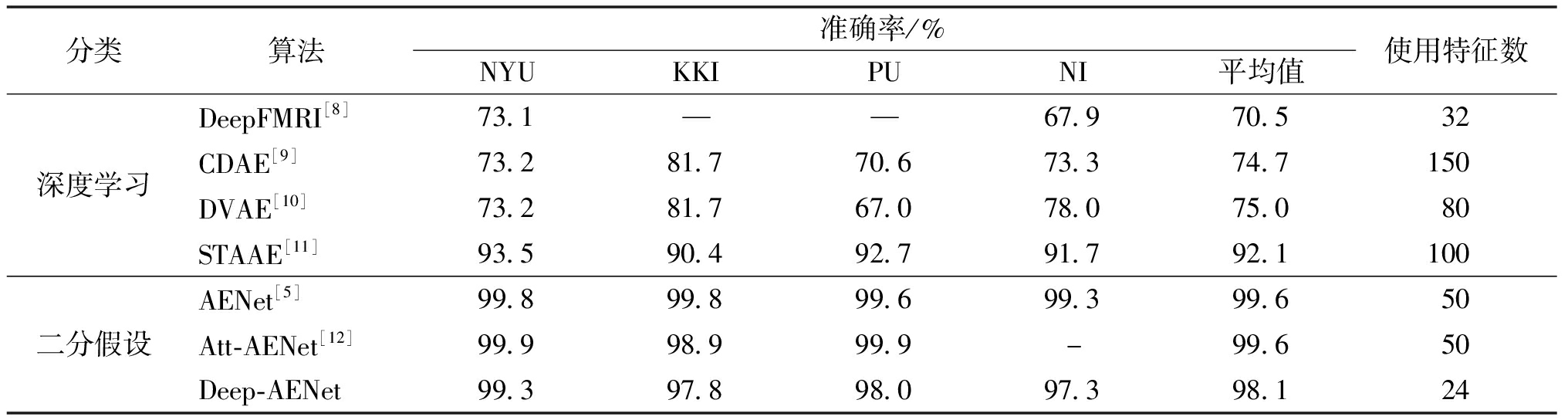
分类算法准确率/%NYUKKIPUNI平均值使用特征数深度学习DeepFMRI[8]73.1——67.970.532CDAE[9]73.281.770.673.374.7150DVAE[10]73.281.767.078.075.080STAAE[11]93.590.492.791.792.1100二分假设AENet[5]99.899.899.699.399.650Att-AENet[12]99.998.999.9-99.650Deep-AENet99.397.898.097.398.124
3.3 生物标识评估
通过注意力编码子网络中的注意力分数的计算和统计进行生物标识的识别与分析。将注意力分数进行排序,边缘系统相关区域的注意力分数排序如图4所示。各子数据库的注意力分数分别在统计20次后求得均值,考虑到数据库大小和准确率的影响,最终每个脑区上的注意力分数计算公式为
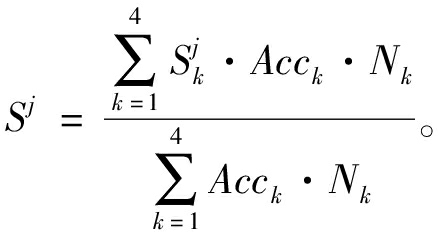
(8)
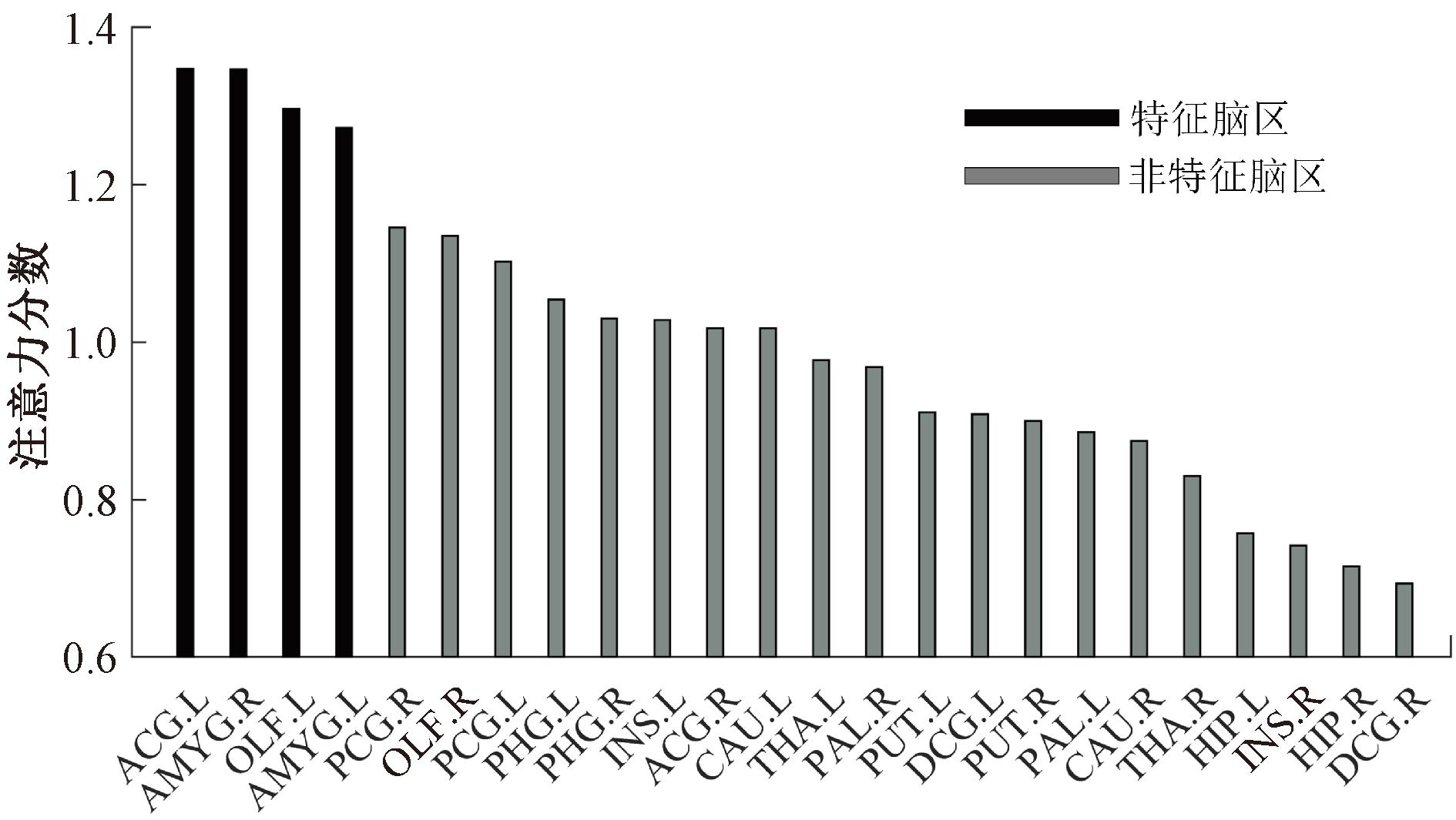
图4 注意力分数排序
Figure 4 Attention score ranking
式中:Sj为j脑区上的平均注意力分数;![]() 为第k个子数据库上j脑区的注意力分数;Acck为第k个子数据库上的准确率;Nk为第k个子数据库的样本量。
为第k个子数据库上j脑区的注意力分数;Acck为第k个子数据库上的准确率;Nk为第k个子数据库的样本量。
前扣带与旁扣带脑回(anterior cingulate and paracingulate gyri,ACG.L)、右杏仁核(amygdala,AMYG.R)、嗅皮质(olfactory cortex,OLF.L)和左杏仁核(amygdala,AMYG.L)是经过生物标识评估得出的典型脑区,因此作为ADHD 生物标识,这可以在ADHD的研究中得到佐证。上述生物标识在大脑中的位置如图5所示。
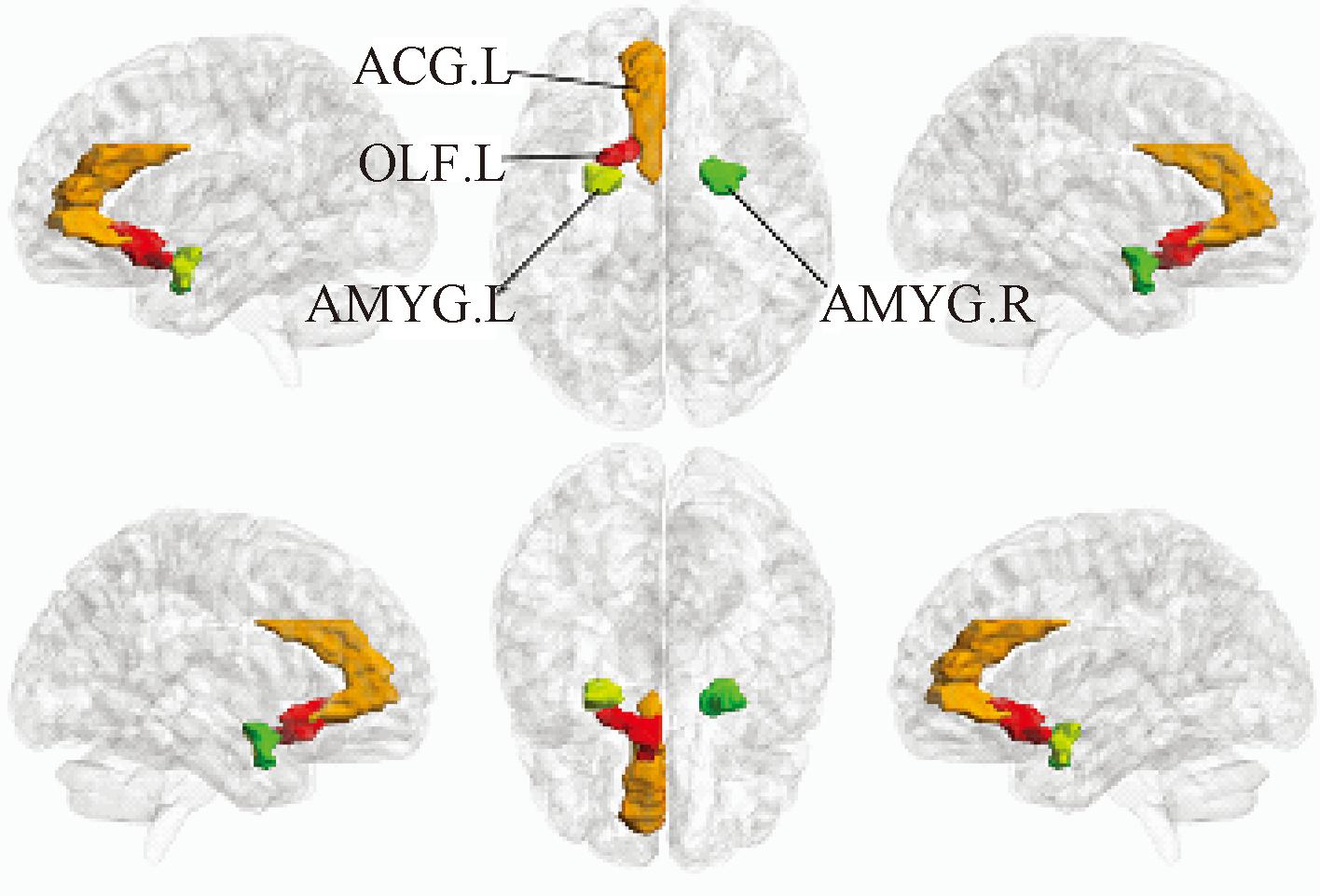
图5 ADHD生物标识的可视化
Figure 5 Visualization of ADHD biomarkers
3.4 生物标识分析
对于上述生物标识,本文进行了线性分析,结果如表6所示。分析过程使用归一化后的ALFF值和症状分数(来自ADHD-200数据库中的表型文件)来进行。另外,根据表型文件排除了共病受试者的数据,以减少其他疾病对相关性结果的影响。由表6可知,PU数据库上在置信区间设置为95%的情况下,相关性值基本都高于0.2,4个生物标识与症状分数均有着明显的相关性,这进一步表明了所得生物标识的正确性。PU数据库上ALFF值与ADHD症状分数的关系,如图6所示。
表6 子数据库上生物标识与症状分数的相关性分析
Table 6 Detected biomarkers and their correlations with symptom scores on sub-database
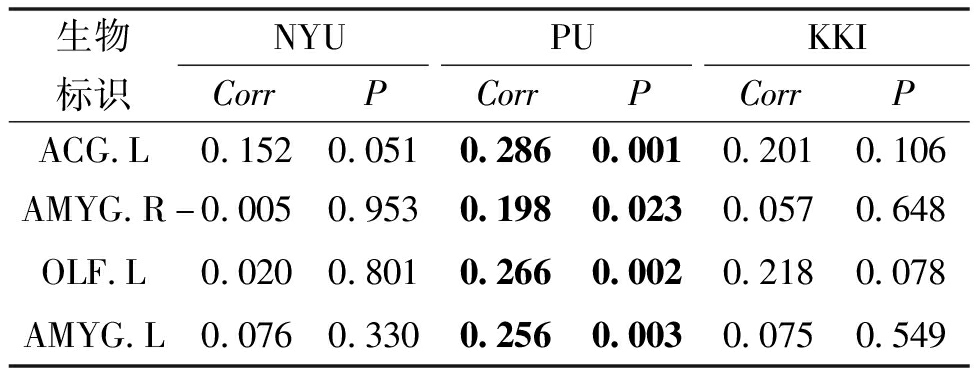
生物标识NYUPUKKICorrPCorrPCorrPACG.L0.1520.0510.2860.0010.2010.106AMYG.R-0.0050.9530.1980.0230.0570.648OLF.L0.0200.8010.2660.0020.2180.078AMYG.L0.0760.3300.2560.0030.0750.549
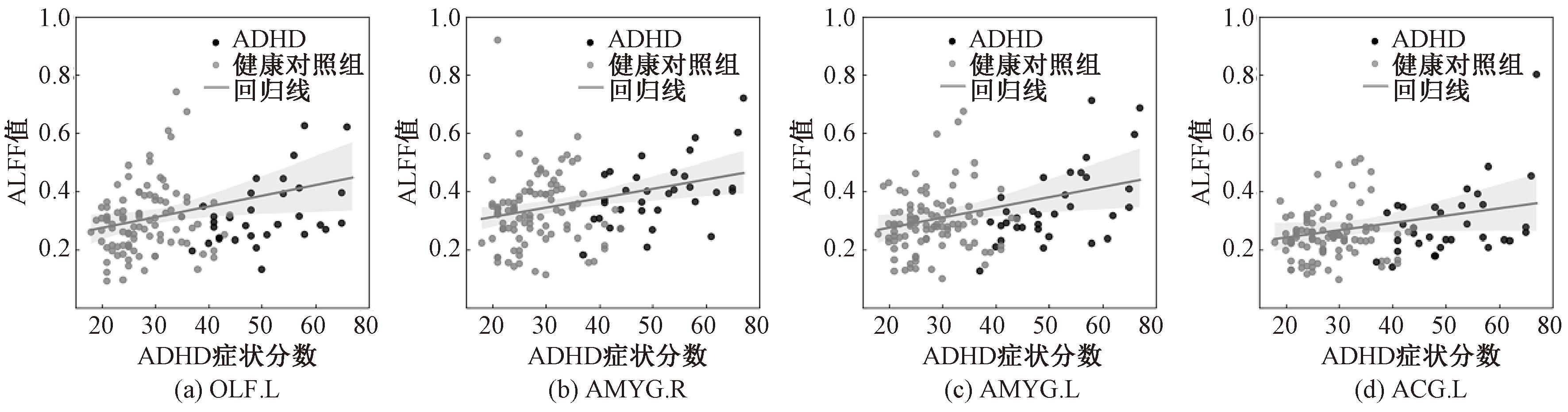
图6 PU子数据库上各生物标识与症状分数的相关性分析
Figure 6 Correlation analysis between biomarkers and symptom scores on PU sub-database
4 结论
本文提出一种端到端深度学习网络,结合二分假设框架,探索边缘系统上ALFF数据在ADHD研究中的分类准确率,以及边缘系统对ADHD的影响。在研究过程中,引入注意力机制,加强网络对高贡献数据的学习,高效地提取ADHD特征,同时用来检测生物标识。实现端到端的特征学习,避免深度学习和机器学习结合带来的特征数目设定问题,提高了特征提取的效率和准确率。实验表明,本文方法优于当前大部分ADHD分类方法,在留一交叉验证中,ADHD分类平均准确率达到98.1%。生物标识的注意力分数统计显示,前扣带与旁扣带脑回、右杏仁核、嗅皮质和左杏仁核是对ADHD疾病影响最显著的4个脑区。本文通过二分假设端到端深度学习框架的应用,实现了边缘系统相关ALFF数据的ADHD分类高准确率,探索了ALFF数据表征ADHD疾病特征的能力和边缘系统对ADHD的影响,证实了二分假设端到端模型的合理性。
[1] LEFFA D T,CAYE A,ROHDE L A.ADHD in children and adults:diagnosis and prognosis[J]Current Topics in Behavioral Neurosciences,2022,57:1-18.
[2] POLANCZYK G,JENSEN P.Epidemiologic considerations in attention deficit hyperactivity disorder:a review and update[J].Child and Adolescent Psychiatric Clinics of North America,2008,17(2):245-260.
[3] BARUA P D,DOGAN S,ACHARYA U R.EPSPatNet86:eight-pointed star pattern learning network for detection ADHD disorder using EEG signals[J].Physiological Measurement,2022,43(3):035002.
[4] QUAAK M,VAN DE MORTEL L,THOMAS R M,et al.Deep learning applications for the classification of psychiatric disorders using neuroimaging data:systematic review and meta-analysis[J].NeuroImage Clinical,2021,30:102584.
[5] TANG Y B,SUN J,WANG C,et al.ADHD classification using auto-encoding neural network and binary hypothesis testing[J].Artificial Intelligence in Medicine,2022,123:102209.
[6] SU L H,KAMATA S I.ADHD classification with low-frequency fluctuation feature map based on 3D CBAMe[C]∥2022 7th International Conference on Biomedical Signal and Image Processing (ICBIP).New York:ACM,2022:74-79.
[7] COLBY J B,RUDIE J D,BROWN J A,et al.Insights into multimodal imaging classification of ADHD[J].Frontiers in Systems Neuroscience,2012,6:59.
[8] RIAZ A,ASAD M,ALONSO E,et al.DeepFMRI:end-to-end deep learning for functional connectivity and classification of ADHD using fMRI[J].Journal of Neuroscience Methods,2020,335:108506.
[9] LIU S Q,ZHAO L,ZHAO J,et al.Attention deficit/hyperactivity disorder classification based on deep spatio-temporal features of functional magnetic resonance imaging[J].Biomedical Signal Processing and Control,2022,71:103239.
[10] QIANG N,DONG Q L,GE F F,et al.Deep variational autoencoder for mapping functional brain networks[J].IEEE Transactions on Cognitive and Developmental Systems,2021,13(4):841-852.
[11] QIANG N,DONG Q L,LIANG H T,et al.A novel ADHD classification method based on resting state temporal templates (RSTT) using spatiotemporal attention auto-encoder[J].Neural Computing and Applications,2022,34(10):7815-7833.
[12] CHEN Y,GAO Y,JIANG A M,et al.ADHD classification combining biomarker detection with attention auto-encoding neural network[J].Biomedical Signal Processing and Control,2023,84:104733.
[13] 李荪,曹峰.智能语音技术端到端框架模型分析和趋势研究[J].计算机科学,2022,49(增刊1):331-336.LI S,CAO F.Analysis and trend research of end-to-end framework model of intelligent speech technology[J].Computer Science,2022,49(S1):331-336.
[14] BONATH B,TEGELBECKERS J,WILKE M,et al.Regional gray matter volume differences between adolescents with ADHD and typically developing controls:further evidence for anterior cingulate involvement[J].Journal of Attention Disorders,2018,22(7):627-638.
[15] UGARTE G,PI A R,CONTRERAS D,et al.Attention deficit-hyperactivity disorder (ADHD):from abnormal behavior to impairment in synaptic plasticity[J].Biology,2023,12(9):1241.
A R,CONTRERAS D,et al.Attention deficit-hyperactivity disorder (ADHD):from abnormal behavior to impairment in synaptic plasticity[J].Biology,2023,12(9):1241.
[16] TANG Y B,WANG C,CHEN Y,et al.Identifying ADHD individuals from resting-state functional connectivity using subspace clustering and binary hypothesis testing[J].Journal of Attention Disorders,2021,25(5):736-748.
[17] TANG Y B,LI X F,CHEN Y,et al.High-accuracy classification of attention deficit hyperactivity disorder with l2,1-norm linear discriminant analysis and binary hypothesis testing[J].IEEE Access,2020,8:56228-56237.
[18] SMITH S M,JENKINSON M,WOOLRICH M W,et al.Advances in functional and structural MR image analysis and implementation as FSL[J].NeuroImage,2004,23(S1):S208-S219.
[19] BELLEC P,CHU C,CHOUINARD-DECORTE F,et al.The Neuro Bureau ADHD-200 Preprocessed repository[J].NeuroImage,2017,144(Pt B):275-286.
[20] LI X,YU R Q,HUANG Q,et al.Alteration of whole brain ALFF/fALFF and degree centrality in adolescents with depression and suicidal ideation after electroconvulsive therapy:a resting-state fMRI study[J].Frontiers in Human Neuroscience,2021,15:762343.
[21] HU B,YU Y,WANG W,et al.MICA:a toolkit for multimodal image coupling analysis[J].Journal of Neuroscience Methods,2021,347:108962.