脑胶质瘤是一种具有高发病率和高致死率的原发性脑肿瘤,对人体的健康造成极大的危害。对脑胶质瘤核磁共振图像的分割可以帮助医生观察和分析脑胶质瘤的外部形态,从而进行诊断治疗。近年来,随着深度学习的发展,以U-Net[1]为主的全卷积神经网络在医学影像分割任务中占据主导地位。U-Net通过构建具有跳跃连接的非对称编码器-解码器结构,达到了良好的医学影像分割效果。其中,编码器由一系列卷积层和下采样层组成,用于提取深层次特征;解码器将深层次特征逐步进行上采样,另外,解码器通过跳跃连接与编码器不同尺寸的特征进行融合,以补充下采样过程中带来的空间信息丢失。此后,基于 U-Net的一系列网络也在医学图像分割领域得到应用,如Res-UNet[2]、V-Net[3]等。
然而,缺乏长距离依赖关系捕捉能力使得卷积神经网络并不能满足工业界的分割精度要求。尽管一些工作采用了空洞卷积[4-5]来克服这一缺陷,但仍然存在局限性。近年来,Transformer[6]在计算机视觉领域取得突破性成就,开始被应用于医学影像分割领域,如TransUNet[7]、AA-TransUNet[8]、TransBTS[9]等。TransUNet[7]首次探索了Transformer在医学图像分割领域的可行性,其总体架构遵循U-Net的设计,利用Transformer将来自卷积神经网络的特征图编码为提取全局上下文的输入序列。同样,TransBTS[9]将Transformer用于编码器末端进行全局信息建模,实现了良好的脑胶质瘤图像分割效果。但以上方法并未考虑大尺寸特征图下的长距离依赖关系。另外,其采用的Transformer自注意力机制耗费内存且计算量大。
为了解决自注意力机制内存和计算量消耗过大的问题,研究者们设计了不同的稀疏自注意力机制。PVT[10]通过空间减少注意力(spatial-reduction attention)降低计算量,Swin Transformer[11]则是限制在一个窗口中计算自注意力,以此降低计算量。很快,这2种方法也被应用于医学影像分割任务中,如Swin-UNet[12]、UTNet[13]等。但是,这2种方法中自注意力机制的设计可能会丢失关键信息,限制自注意力机制建立长距离关系依赖的能力。最近,具有可变形注意力的Transformer[14]通过设计一种名为可变形注意力的稀疏自注意力机制来缓解这一缺陷,并取得了更好的效果。
此外,Transformer的自注意力机制缺乏局部上下文信息提取能力,为了解决上述问题,CoAtNet[15]、CMT[16]等将卷积引入Transformer模型中,增强视觉Transformer的局部性,从而获得了更优的性能,这也验证了CNN与Transformer混合方法的有效性。
受上述研究的启发,本文提出一种基于CNN-Transformer混合的脑胶质瘤图像分割方法(Med-CaDA)。不同于TransUNet、TransBTS仅仅将Transformer应用于小尺寸特征图,本文采用了稀疏自注意力机制,并将其应用于各个尺寸特征图中提取全局上下文信息,建立不同分辨率下局部和全局的依赖关系。此外,将卷积的瓶颈残差模块和可变形注意力Transformer组成串行模块,命名为CaDA块,该模块既保留了卷积局部上下文提取的优势,又借助了Transformer全局信息捕捉的能力。
1 基于卷积和可变形注意力的脑胶质瘤图像分割方法
图1为Med-CaDA整体架构和CaDA块。Med-CaDA的整体架构如图1(a)所示,其遵循U-Net的编码器-解码器架构设计,由6个部分组成:嵌入层、CaDA块、下采样层、上采样层、扩展层和跳跃连接。输入为X∈RH×W×D×K,其中H、W、D和K分别表示空间分辨率的高度和宽度、切片深度和模态数量。
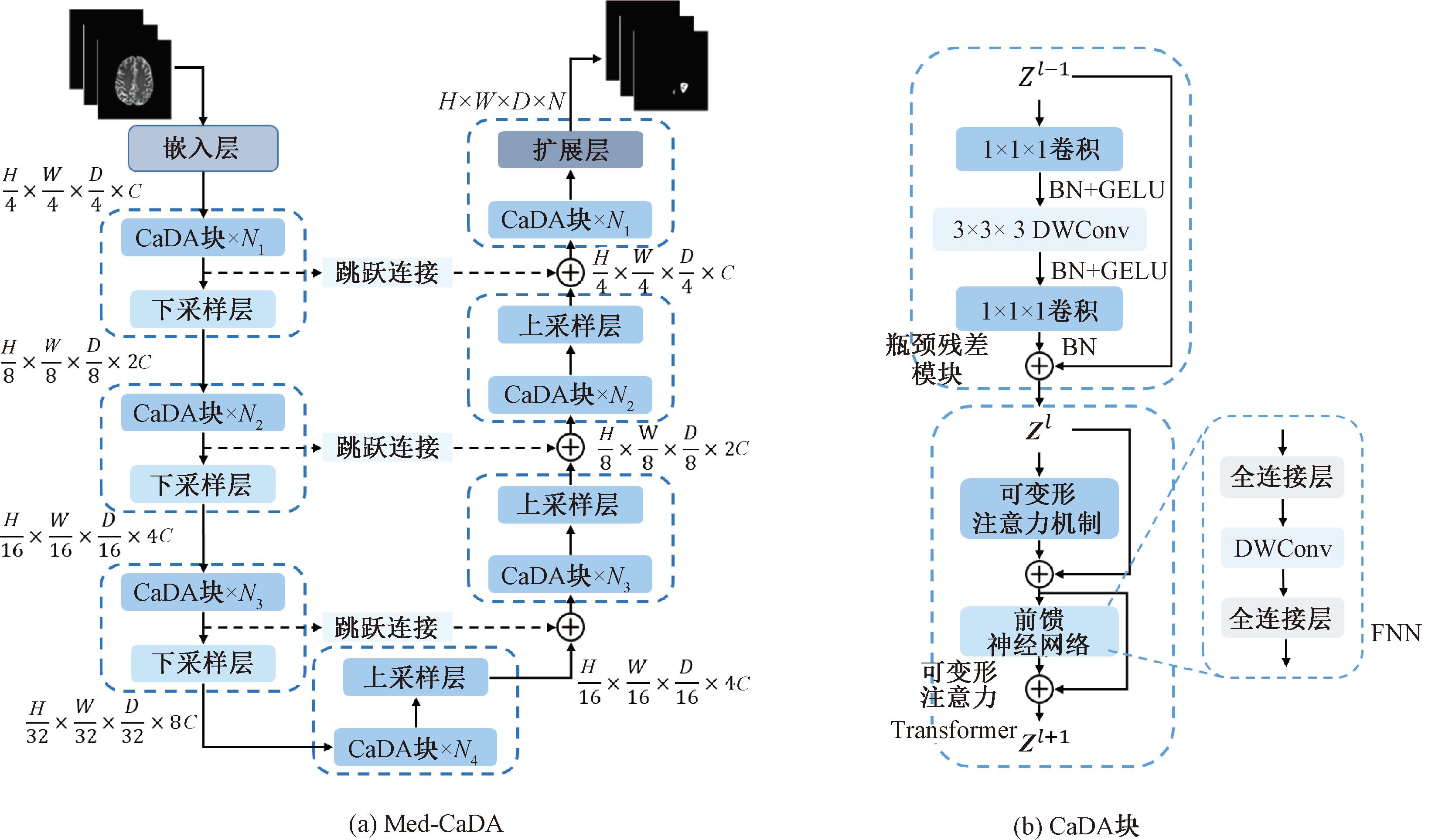
图1 Med-CaDA整体架构和CaDA块
Figure 1 Overall architecture of Med-CaDA and CaDA block
1.1 嵌入层和扩展层
嵌入层和扩展层如图2(a)、2(c)所示,图中GELU表示激活函数,LN表示LayerNorm标准化。嵌入层由2个步长为2的3×3×3卷积组成,逐步将输入图像X∈RH×W×D×K映射为特征向量![]() 用于更精确地编码图像的像素级空间信息。
用于更精确地编码图像的像素级空间信息。
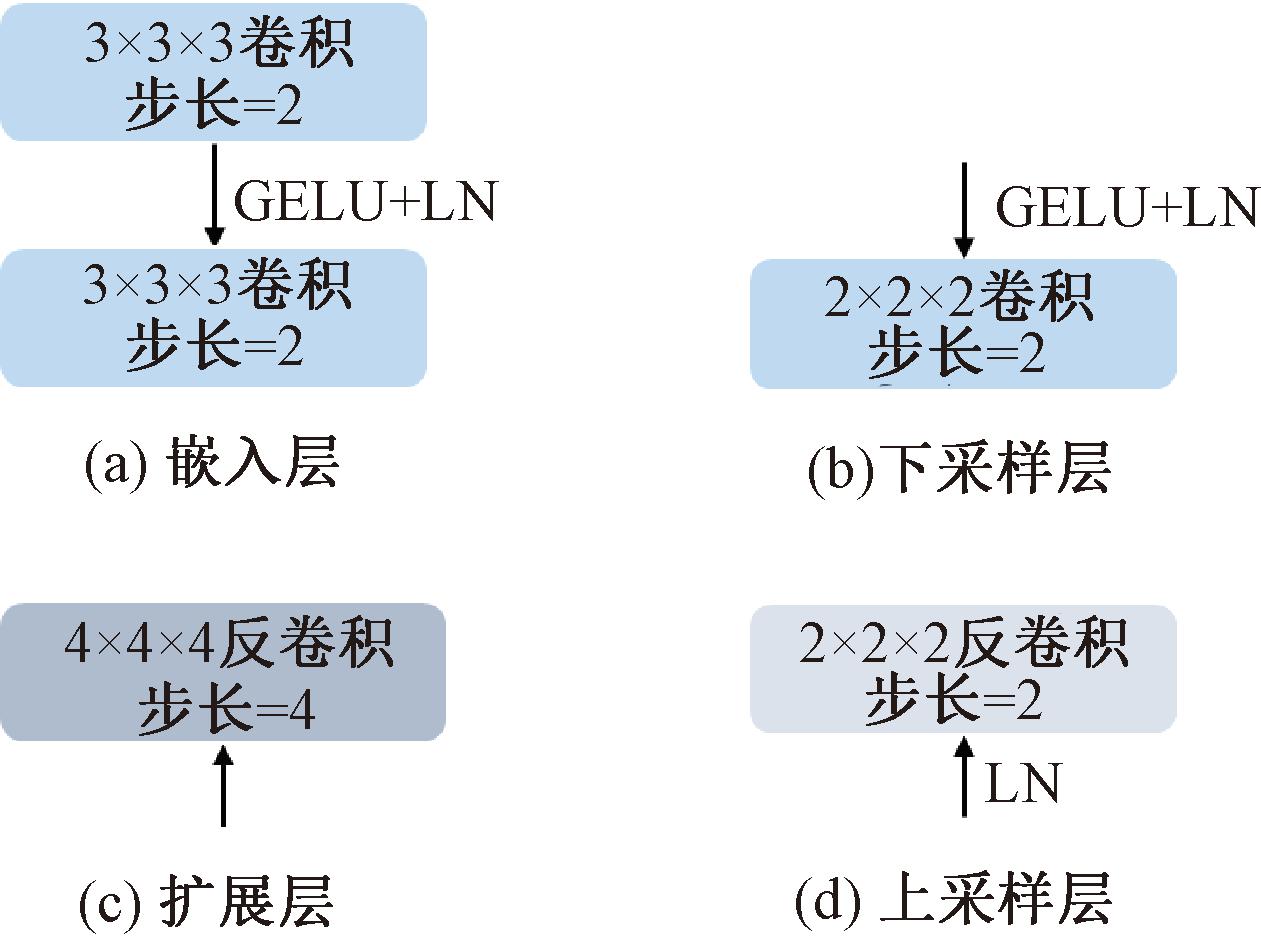
图2 嵌入层、扩展层、下采样层和上采样层示意图
Figure 2 Schematic diagram of embedding, expanding, down-sampling and up-sampling layer
与嵌入层相对应,扩展层是一个步长为4的4×4×4卷积,负责将高维度张量还原回输入图像尺寸Y∈RH×W×D×N,其中N表示分割类别数量。
1.2 下采样层和上采样层
下采样层和上采样层是构建编码器-解码器分层架构的关键,如图2(b)、2(d)所示,下采样层采用步长为2的2×2×2卷积,逐步将特征图编码为尺寸更小、维度更高的深层特征。上采样层与下采样层对应,是一个步长为2的2×2×2反卷积,用于将深层特征图尺寸加倍,维度减半。
1.3 CaDA块
卷积和自注意力机制分别擅长局部上下文信息提取和长距离依赖捕捉,两者对于脑胶质瘤图像分割这类密集预测任务都至关重要。因此,本文设计了由卷积的瓶颈残差模块和可变形注意力Transformer串行组成的CaDA块,如图1(b)所示,图中BN、GELU、DWConv分别表示BatchNorm标准化、激活函数和深度可分离卷积。
(1)瓶颈残差模块。图1(b)上半部分是瓶颈残差模块,依次由1×1×1卷积、3×3×3深度可分离卷积、1×1×1卷积构成,采用深度可分离卷积可大幅度降低计算量和参数量。另外,不同于MobileNetV2[17]中2个1×1×1卷积用于先升维后降维,本文则先降维再升维,从而在计算3×3×3卷积时进一步降低计算量。计算过程可以表述为
Zl=Bottleneck(Zl-1)+Zl-1;
(1)
Bottleneck(X)=Conv(DWConv(Conv(X)))。
(2)
式中:Conv(·)和DWConv(·)分别表示1×1×1卷积和深度可分离卷积。
(2)可变形注意力Transformer。图1(b)下半部分显示的是可变形注意力Transformer,由可变形注意力机制、前馈神经网络FNN和残差连接组成。受DAT[14]启发,本文实现了三维数据下的可变形注意力机制,如图3所示。可变形注意力机制的键值向量和值向量是在原图上采样特征投影得到的,这些采样特征由查询向量通过一个偏置学习网络学习的采样点经过双线性插值得到。具体实现过程如下。
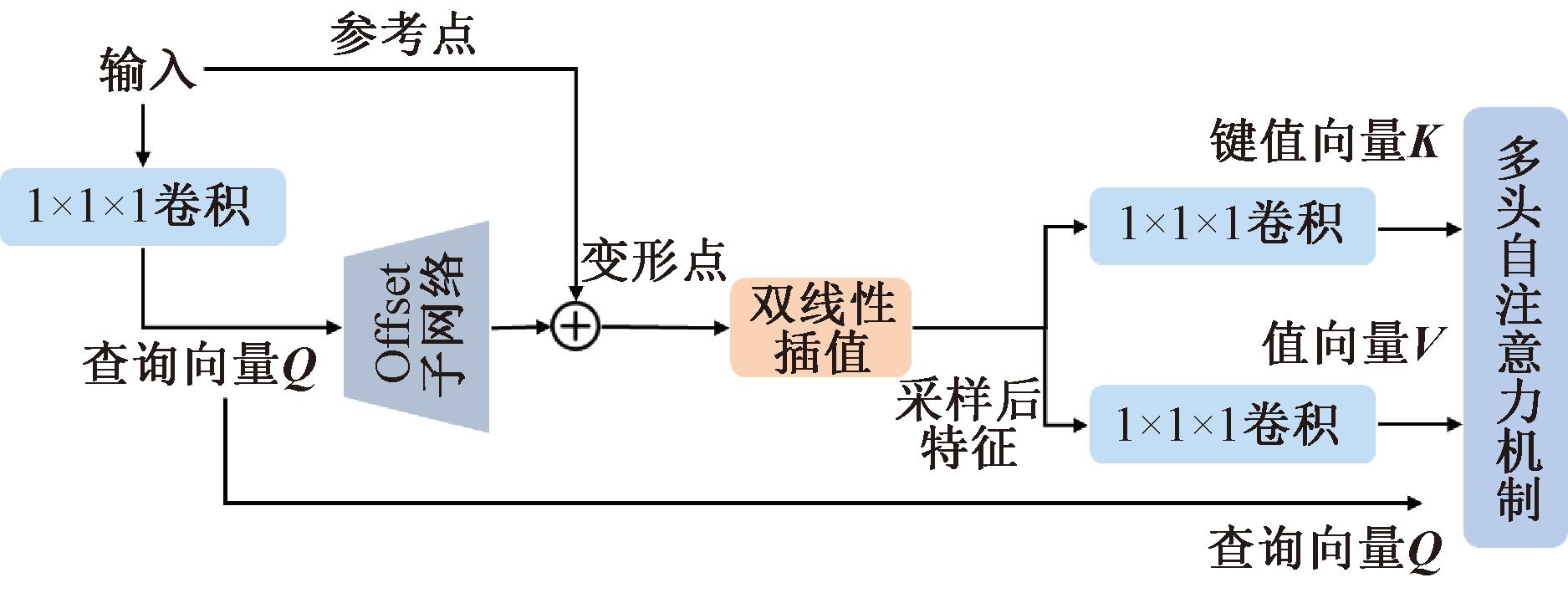
图3 可变形注意力机制示意图
Figure 3 Schematic diagram of deformable attention
假设给定输入X∈RH×W×D×C,首先对输入投影产生查询向量Q,同时根据输入X的尺寸生成一个大小为![]() 的均匀网格点(F为预定义参数),称之为参考点,参考点的值是线性间隔的三维坐标
的均匀网格点(F为预定义参数),称之为参考点,参考点的值是线性间隔的三维坐标![]() 然后对参考点的值归一化到[-1,1],那么三维坐标将变成{(-1,-1,-1),…,(1,1,1)}。随后,将查询向量Q输入到偏置学习子网络Offset(·)以生成每个参考点的偏移量ΔP(为了防止偏移量过大,采用可变参数s和tanh(·)去控制),然后通过参考点和偏移量ΔP相加得到变形点,根据变形点所对应的坐标位置在输入上采用双线性插值采样得到采样后的特征图Xz,随后投影产生键值向量K和值向量V。最后,对得到的Q、K和V计算多头自注意力输出结果。可变形多头注意力机制(deformable multi-head attention,DMHA)计算公式为
然后对参考点的值归一化到[-1,1],那么三维坐标将变成{(-1,-1,-1),…,(1,1,1)}。随后,将查询向量Q输入到偏置学习子网络Offset(·)以生成每个参考点的偏移量ΔP(为了防止偏移量过大,采用可变参数s和tanh(·)去控制),然后通过参考点和偏移量ΔP相加得到变形点,根据变形点所对应的坐标位置在输入上采用双线性插值采样得到采样后的特征图Xz,随后投影产生键值向量K和值向量V。最后,对得到的Q、K和V计算多头自注意力输出结果。可变形多头注意力机制(deformable multi-head attention,DMHA)计算公式为
Q=XWq;
(3)
ΔP=s·tanh(Offset(Q));
(4)
Xz=BI(X,P+ΔP);
(5)
K=XzWk;
(6)
V=XzWv;
(7)
(8)
DMHA(Z)=Concat(Z(1),Z(2),…,Z(m))Wo。
(9)
式中:Offset(·)是依次由1×1×1卷积、深度可分离卷积和1×1×1卷积组成的子网络;![]() 表示参考点的偏移量;BI(·)表示双线性插值;Wq∈RC×C、Wk∈RC×C、Wv∈RC×C分别表示查询向量Q∈RN×C、键值向量K∈RM×C和值向量V∈RM×C的投影矩阵,其中N=H×W×D,M=H/F×W/F×D/F;Q(m)∈RN×d,K(m)、V(m)∈RM×d分别代表第m个头部下的3个向量;
表示参考点的偏移量;BI(·)表示双线性插值;Wq∈RC×C、Wk∈RC×C、Wv∈RC×C分别表示查询向量Q∈RN×C、键值向量K∈RM×C和值向量V∈RM×C的投影矩阵,其中N=H×W×D,M=H/F×W/F×D/F;Q(m)∈RN×d,K(m)、V(m)∈RM×d分别代表第m个头部下的3个向量;![]() 代表每一个头部的维度,h为头部数量;Z(m)∈RN×d为第m个头部的注意力输出;Wo∈RC×C是可变形多头注意力机制的输出投影矩阵。
代表每一个头部的维度,h为头部数量;Z(m)∈RN×d为第m个头部的注意力输出;Wo∈RC×C是可变形多头注意力机制的输出投影矩阵。
另外,前馈神经网络(feedforward neural network,FNN)由2个全连接层和1个深度可分离卷积组成,如图1(b)所示。将深度可分离卷积引入前馈网络中可以为Transformer模块增加局部性。
最终,结合可变形多头注意力、前馈神经网络和残差连接,可变形注意力Transformer计算公式可以表示为
(10)
(11)
式中:DMHA(·)表示可变形多头注意力机制;LN(·)表示LayerNorm标准化;FNN(·)表示前馈神经网络。
2 实验与分析
2.1 数据集构成
本文采用BraTS2020脑胶质瘤图像分割数据集,训练集和验证集分别由369个和125个3维 MRI组成,每个MRI包括4种模态:T1、T1ce、Flair和T2。需要分割的3个类别分别为整个肿瘤区域(whole tumor,WT)、肿瘤核心区域(tumor core,TC)以及活动肿瘤区域(enhance tumor,ET)。本文在训练集上进行训练,在验证集上进行测试,并将验证集上的预测标签上传到BraTS2020比赛官网以得到分割结果。
2.2 实验环境和参数设置
实验采用的编程语言为Python 3.8,深度学习框架为Pytorch 1.7.1,使用的显卡为2张Tesla T4,显存一共为32 GB。实验中,设置Med-CaDA模型不同阶段预定义参数如表1所示,预定义参数包括每阶段的分辨率、循环次数N、通道数C、可变形注意力的预定义参数F和s、多头注意力机制的头数h。在训练过程中,沿用了TransBTS[9]的随机裁剪、随机镜像翻转和随机强度偏移3种数据增强策略。采用Adam优化器,学习率设置为1×10-4,batch size为4,训练轮数为800。另外,采用余弦学习率衰减策略控制学习率大小,便于模型收敛;采用L2正则化去缓解模型过拟合问题(权重衰减设置为1×10-5)。
表1 不同阶段预定义参数设置
Table 1 Predefined parameters setting at different stages

阶段分辨率相关系数阶段一32×32×32N=1,C=96,F=4,s=-1,h=3阶段二16×16×16N=1,C=192,F=2,s=-1,h=6阶段三8×8×8N=2,C=384,F=1,s=2,h=12阶段四4×4×4N=1,C=768,F=1,s=2,h=24
2.3 评估指标
在脑胶质瘤图像分割实验中采用了浮点运算次数衡量模型的复杂度。采用Dice系数和95%豪斯多夫距离2个评估指标衡量2个点集合间的相似程度,Dice系数对集合内部填充比较敏感,95%豪斯多夫距离对边界比较敏感。可以表示为
(12)
HD(A,B)=max(h(A,B),h(B,A));
(13)
(14)
(15)
式中:|A∩B|表示A、B间交集的元素个数;|A|和|B|分别表示A和B的元素个数;h(A,B)的实际意义为计算集合B到集合A每个点距离最近的距离并排序,然后选择距离中的最大值。
2.4 实验结果分析
2.4.1 推理实验
损失函数用来估量模型的预测值与真实值不一致的程度,损失函数越小,模型效果就越好。训练过程中损失函数变化曲线如图4所示。由图4可知,损失值随着训练轮数的增加逐步减少,逐渐趋近于0,说明预测值越来越接近于真实值,模型的性能越来越好,进而说明将该模型用于BraTS2020数据集的分割是有效的。
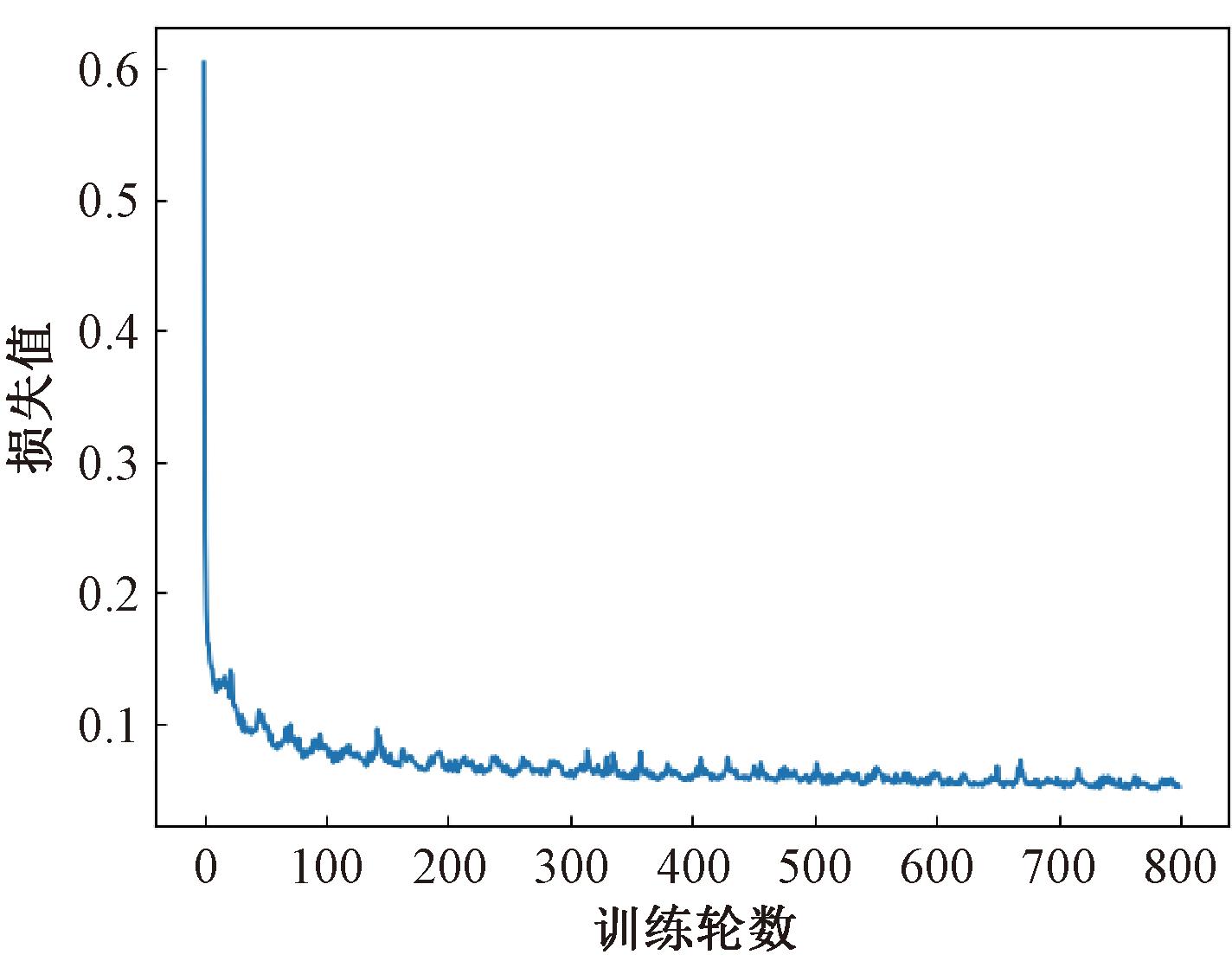
图4 损失函数变化曲线
Figure 4 Curve of loss function
另外,为了验证所采用的可变形注意力机制的有效性,采用了几种不同的稀疏自注意力机制并进行实验,包括空间减少注意力[10](SRA)、窗口注意力[11](WA)以及可变形注意力(DA)。实验结果如表2所示。由表2可知,采用可变形注意力在Dice系数指标上完全优于其他的注意力机制,在95%豪斯多夫距离指标上ET和TC的效果也优于其他注意力机制。由此表明,可变形注意力以数据依赖的方式选择键值向量和值向量,更有助于建立长距离依赖关系。
表2 不同稀疏注意力机制在BraTS2020数据集中的结果
Table 2 Results of the BraTS2020 dataset under different sparse self-attention
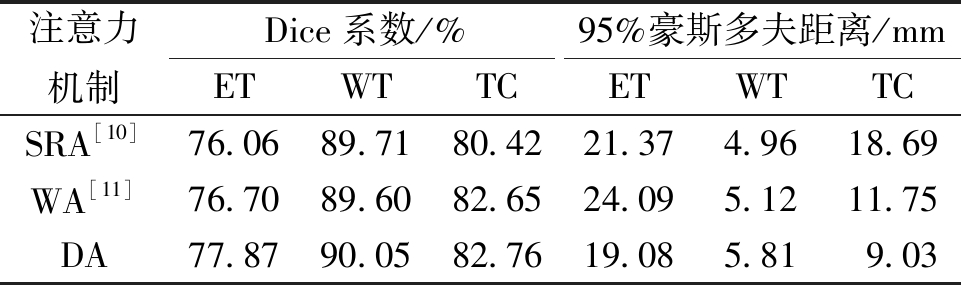
注意力机制Dice系数/%95%豪斯多夫距离/mmETWTTCETWTTCSRA[10]76.0689.7180.4221.374.9618.69WA[11]76.7089.6082.6524.095.1211.75DA77.8790.0582.7619.085.819.03
2.4.2 对比实验
为了验证Med-CaDA在脑胶质瘤分割的有效性,在BraTS2020脑胶质瘤图像分割数据集上进行对比实验。选择了3种基于卷积的经典方法、2种基于卷积的先进方法以及2种引入Transformer的先进方法进行对比,分别为3DU-Net[18]、V-Net[3]、3D Res-UNet[19]、MMTSN[20]、MDNet[21]、TransUNet[7]及TransBTS[9],对比实验结果如表 3所示。
由表3可以看出,Med-CaDA在ET、WT、TC 3个分割指标中,取得的Dice 系数及其平均值分别为77.87%、90.05%、82.76%和83.56%;95%豪斯多夫距离及其平均值分别为19.08、5.81、9.02及11.30 mm。与3个经典的方法相比,Med-CaDA在2个评价指标上均有显著的提升。与2种基于卷积的先进方法和2种引入Transformer的先进方法相比,Med-CaDA在Dice系数指标上虽然只在TC上超过其他方法,但是在平均水平上会高于其他方法。同时,Med-CaDA的单个样本和单个切片下复杂度下降了50%~90%。在95%豪斯多夫距离上虽然未达到最佳,但在平均水平上与最好的方法仅仅相差0.75 mm。所以,Med-CaDA在保证脑胶质瘤图像分割精度的同时,大幅度提高了分割效率。
表3 不同方法的对比实验结果
Table 3 Comparative experimental results of different methods
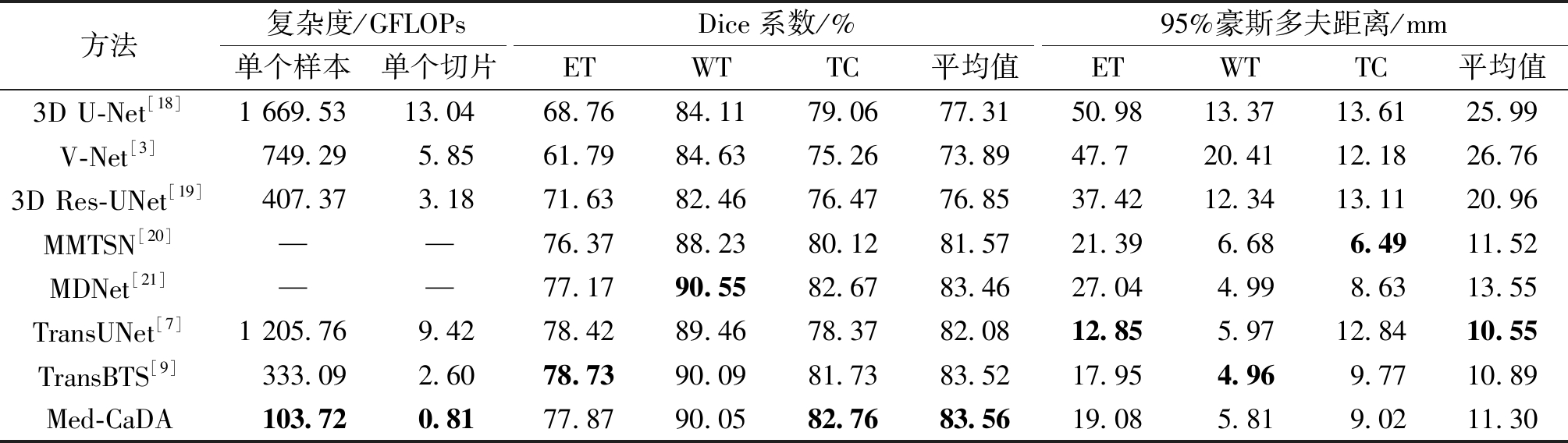
方法复杂度/GFLOPsDice系数/%95%豪斯多夫距离/mm单个样本单个切片ETWTTC平均值ETWTTC平均值3D U-Net[18]1 669.5313.0468.7684.1179.0677.3150.9813.3713.6125.99V-Net[3]749.295.8561.7984.6375.2673.8947.720.4112.1826.763D Res-UNet[19]407.373.1871.6382.4676.4776.8537.4212.3413.1120.96MMTSN[20]——76.3788.2380.1281.5721.396.686.4911.52MDNet[21]——77.1790.5582.6783.4627.044.998.6313.55TransUNet[7]1 205.769.4278.4289.4678.3782.0812.855.9712.8410.55TransBTS[9]333.092.6078.7390.0981.7383.5217.954.969.7710.89Med-CaDA103.720.8177.8790.0582.7683.5619.085.819.0211.30
3 结论
本文提出了一种基于卷积和可变形注意力的脑胶质瘤图像分割方法,该方法继承了卷积建模局部上下文信息的优势,还利用了Transformer学习全局语义相关性,这种CNN-Transformer混合架构可以在没有任何预训练的情况下实现医学影像的精准分割。在BraTS2020数据集上的实验结果表明,与其他方法相比,本文提出的模型在保证分割精度的同时降低了至少50%的计算开销,有效提升了脑胶质瘤图像的分割效率。所以,在脑胶质瘤图像这类医学影像分割任务中,采取稀疏的方法降低参数量同样可以达到良好的分割效果。
[1] RONNEBERGER O, FISCHER P, BROX T. U-Net: convolutional networks for biomedical image segmentation[C]∥International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer, 2015: 234-241.
[2] XIAO X, LIAN S, LUO Z M, et al. Weighted Res-UNet for high-quality retina vessel segmentation[C]∥2018 9th International Conference on Information Technology in Medicine and Education (ITME). Piscataway: IEEE, 2018: 327-331.
[3] MILLETARI F, NAVAB N, AHMADI S A. V-Net: fully convolutional neural networks for volumetric medical image segmentation[C]∥2016 Fourth International Conference on 3D Vision (3DV). Piscataway: IEEE, 2016: 565-571.
[4] FENG S L, ZHAO H M, SHI F, et al. CPFNet: context pyramid fusion network for medical image segmentation[J]. IEEE Transactions on Medical Imaging, 2020, 39(10): 3008-3018.
[5] XIA H Y, MA M J, LI H S, et al. MC-Net: multi-scale context-attention network for medical CT image segmentation[J].Applied Intelligence, 2022, 52(2): 1508-1519.
[6] VASWANI A, SHAZEER N, PARMAR N, et al. Attention is all you need[C]∥Proceedings of the 31st International Conference on Neural Information Processing Systems. New York: ACM, 2017: 6000-6010.
[7] CHEN J N, LU Y Y, YU Q H, et al. AA-TransUNet: transformers make strong encoders for medical image segmentation[EB/OL].(2021-02-08)[2023-01-12]. https:∥doi.org/10.48550/arXiv.2102.04306.
[8] YANG Y, MEHRKANOON S. AA-TransUNet: attention augmented transunet for nowcasting tasks[C]∥2022 International Joint Conference on Neural Networks (IJCNN). Piscataway:IEEE, 2022: 1-8.
[9] WANG W X, CHEN C, DING M, et al. TransBTS: multimodal brain tumor segmentation using transformer[C]∥International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer, 2021: 109-119.
[10] WANG W H, XIE E Z, LI X, et al. Pyramid vision transformer: a versatile backbone for dense prediction without convolutions[C]∥2021 IEEE/CVF International Conference on Computer Vision (ICCV). Piscataway:IEEE, 2022: 548-558.
[11] LIU Z, LIN Y T, CAO Y, et al. Swin Transformer: hierarchical vision transformer using shifted windows[C]∥2021 IEEE/CVF International Conference on Computer Vision (ICCV). Piscataway:IEEE, 2022: 9992-10002.
[12] CAO H, WANG Y, CHEN J, et al. Swin-UNet: Unet-like pure transformer for medical image segmentation[EB/OL]. (2021-05-12)[2023-01-12].https:∥doi.org/10.48550/arXiv.2105.05537.
[13] GAO Y, ZHOU M, METAXAS D N. UTNet: a hybrid transformer architecture for medical image segmentation[C]∥International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer, 2021: 61-71.
[14] XIA Z F, PAN X R, SONG S J, et al. Vision Transformer with deformable attention[C]∥2022 IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR). Piscataway:IEEE, 2022: 4784-4793.
[15] DAI Z H, LIU H X, LE Q V, et al. CoAtNet: Marrying convolution and attention for all data sizes[J]. Advances in Neural Information Processing Systems, 2021, 34: 3965-3977.
[16] GUO J Y, HAN K, WU H, et al. CMT: convolutional neural networks meet vision transformers[C]∥2022 IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR). Piscataway:IEEE, 2022: 12165-12175.
[17] SANDLER M, HOWARD A, ZHU M L, et al. MobileNetV2: inverted residuals and linear bottlenecks[C]∥2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Piscataway:IEEE, 2018: 4510-4520.
[18] ÇIÇEK Ö, ABDULKADIR A, LIENKAMP S S, et al. 3D U-net: learning dense volumetric segmentation from sparse annotation[M]∥Medical Image Computing and Computer-Assisted Intervention-MICCAI 2016. Cham: Springer, 2016: 424-432.
[19] YU W, FANG B, LIU Y Q, et al. Liver vessels segmentation based on 3D residual U-NET[C]∥2019 IEEE International Conference on Image Processing (ICIP).Piscataway: IEEE, 2019: 250-254.
[20] LIU C Y, DING W B, LI L, et al. Brain tumor segmentation network using attention-based fusion and spatial relationship constraint[C]∥International MICCAI Brainlesion Workshop. Cham:Springer, 2021: 219-229.
[21] VU M H, NYHOLM T, LÖFSTEDT T. Multi-decoder networks with multi-denoising inputs for tumor segmentation[C]∥International MICCAI Brainlesion Workshop. Cham: Springer, 2021: 412-423.